각 질병 그룹과 나머지 그룹간의 멀티오믹스 차이를 확인합니다.
위암 그룹 확인
헬빅쇼 데이터셋의 위암 그룹은 "GGG" 입니다. 전체 환자수에서 위암이 차지하는 비율을 파이차트로 확인합니다. 그리고, case 그룹을 위암그룹으로 하고 나머지는 control 로 하여, 비교 분석을 수행합니다.
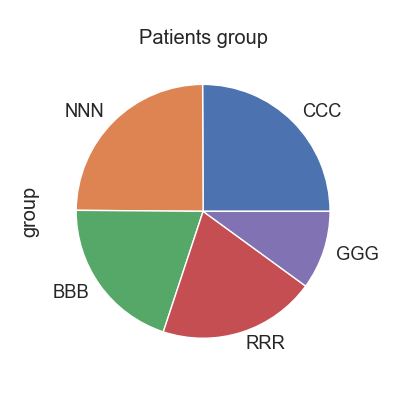
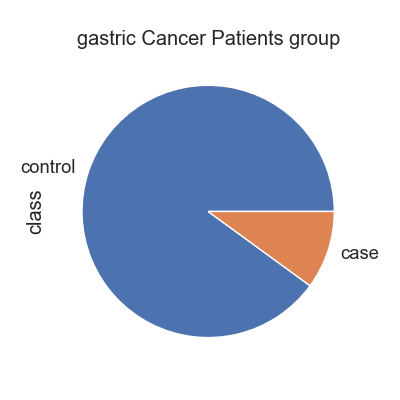
Fig1. 환자 그룹별 파이차트
변이 시그니처 분석
WGS 데이터로 부터, 변이 종류별 변이 갯수를 카운팅한 후, 이를 NMF 분석하여 7개의 시그니처를 생성합니다. case, control 각각 시그니처 분포를 확인합니다.
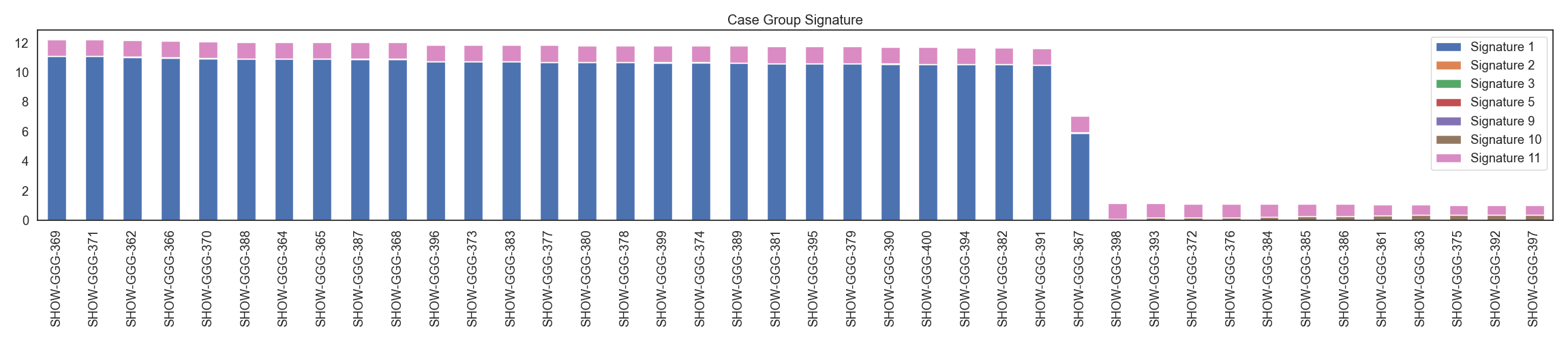
Fig2. Case 그룹 시그니처 분포
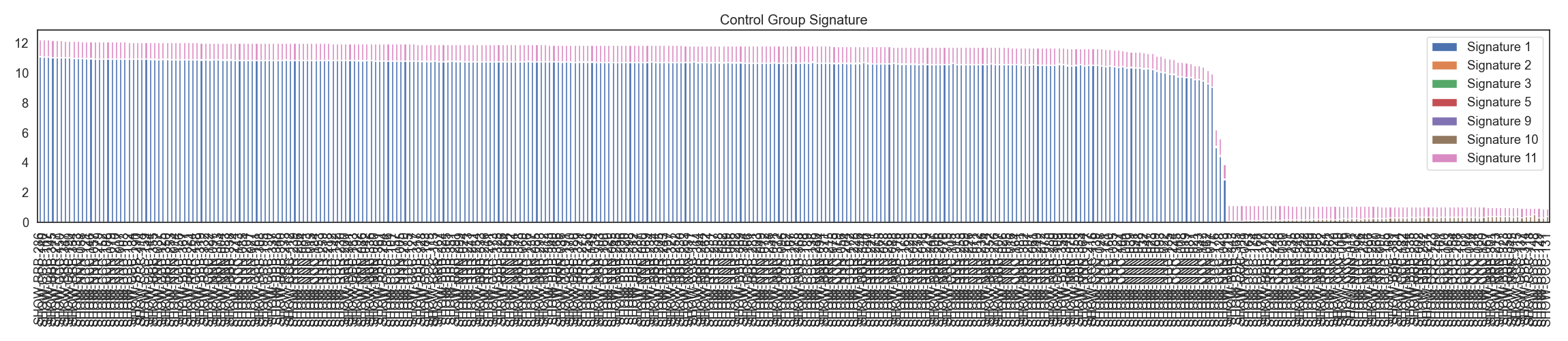
Fig3. Control 그룹 시그니처 분포
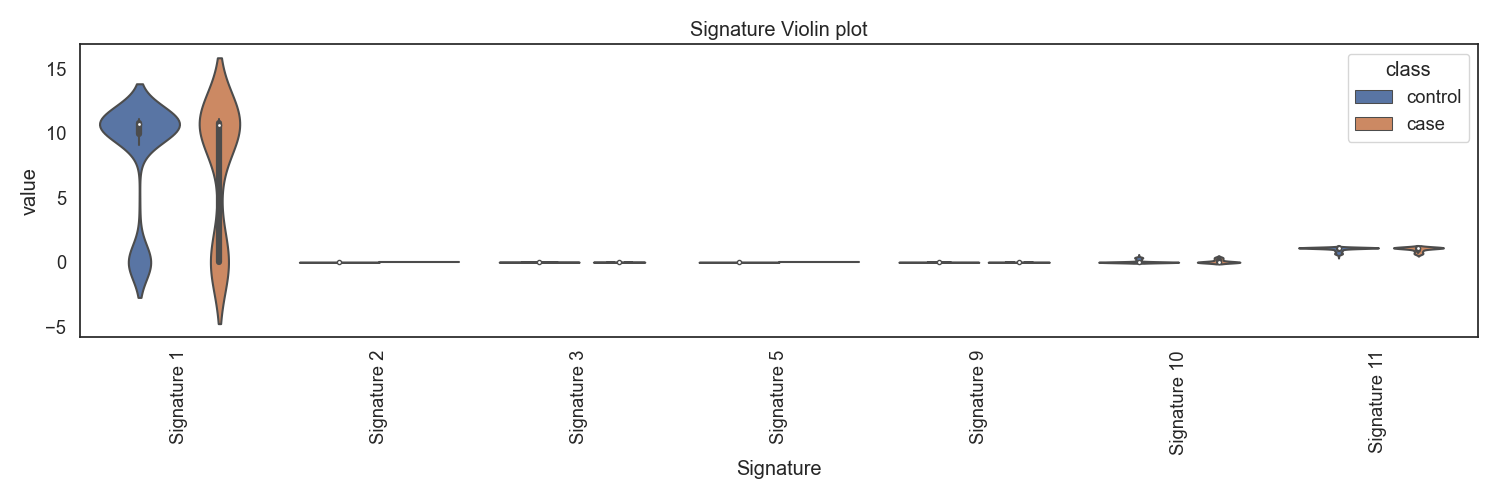
Fig4. 시그니처별 바이올린 플랏
DNA 메틸화 비율
Methyl-seq 데이터로 부터, 유전자별 DNA 메틸화 비율을 계산하고, 환자별로 평균을 계산한 후, 비교합니다.
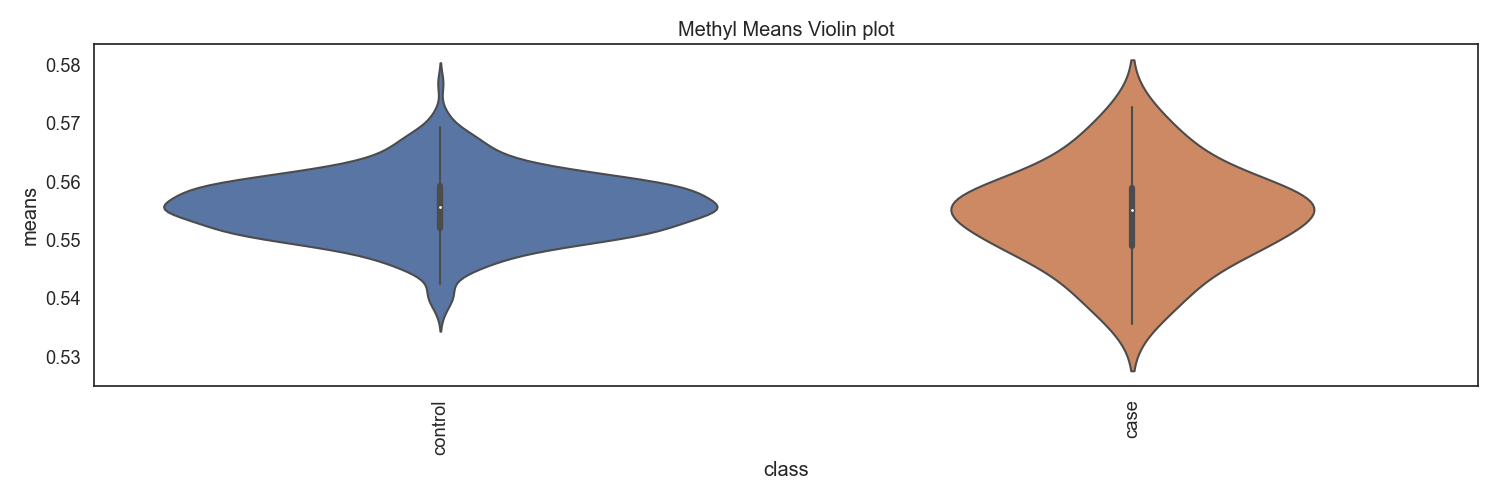
Fig5. 클래스별 메틸화 바이올린 플랏
Case/control 그룹간 차이가 큰 유전자 상위 10개의 유전자목록은 다음과 같습니다.
Fig6. 메틸화의 차가 양으로 큰 gene
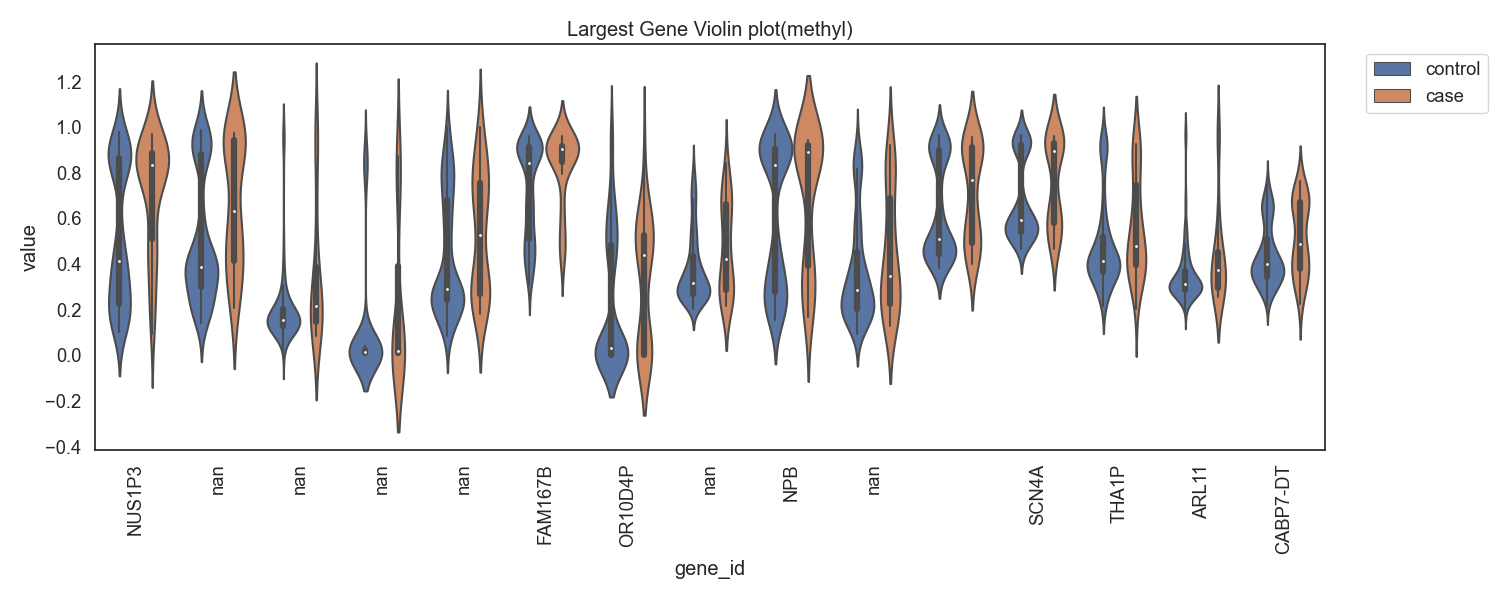
Fig7. 메틸화의 차가 양으로 큰 gene 바이올린 플랏
Fig8. 메틸화의 차가 음으로 큰 gene
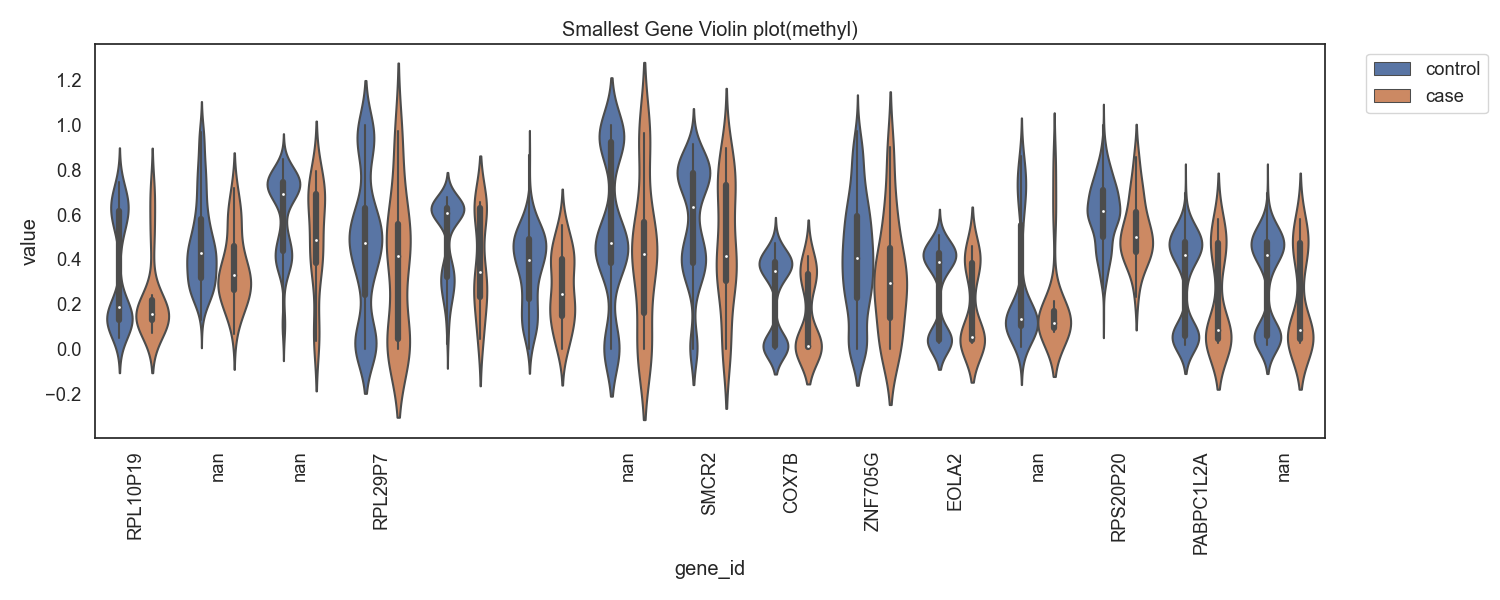
Fig9. 메틸화의 차가 음으로 큰 gene 바이올린 플랏
유전자 발현 분석
RNA-seq 분석을 통해 확인한 TPM 자료로 부터, Case/control 그룹간 전체 환자별 유전자 발현 평균 분포를 확인합니다.
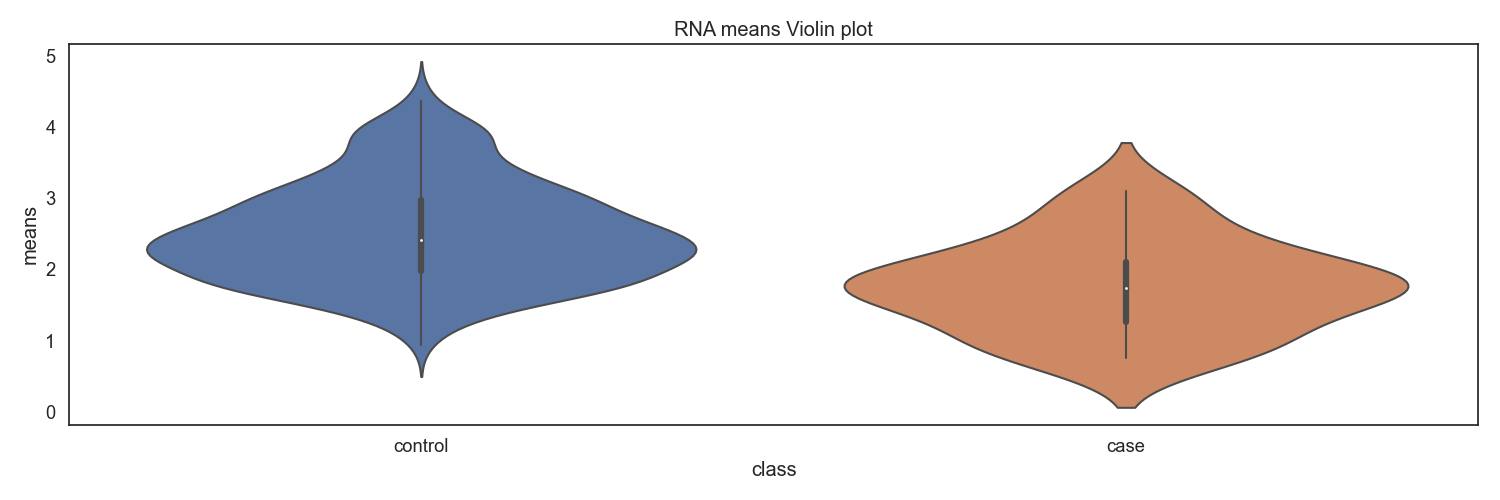
Fig10. 클래스별 RNA발현량 바이올린 플랏
Case/control 그룹간 유전자 발현 차이가 높은 유전자들의 목록을 확인합니다.
Fig11. RNA발현량의 차가 음으로 큰 gene
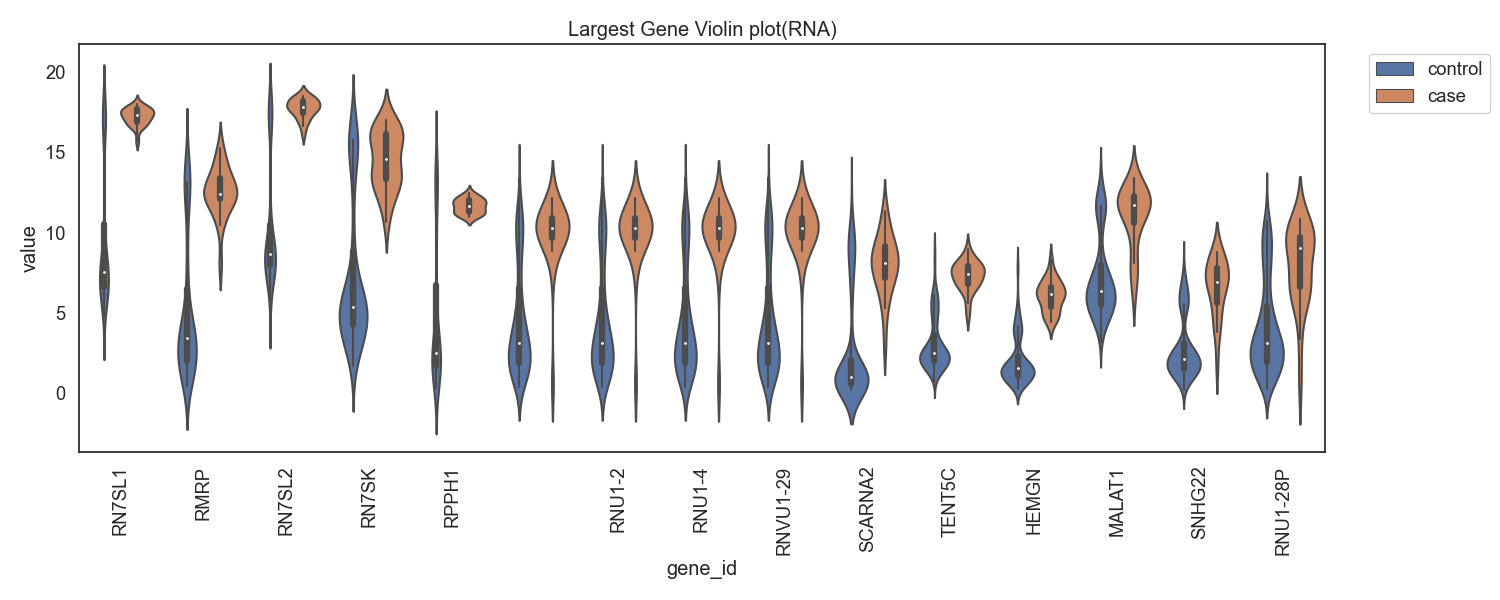
Fig12. RNA발현량의 차가 양으로 큰 gene 바이올린 플랏
Fig13. RNA발현량의 차가 음으로 큰 gene
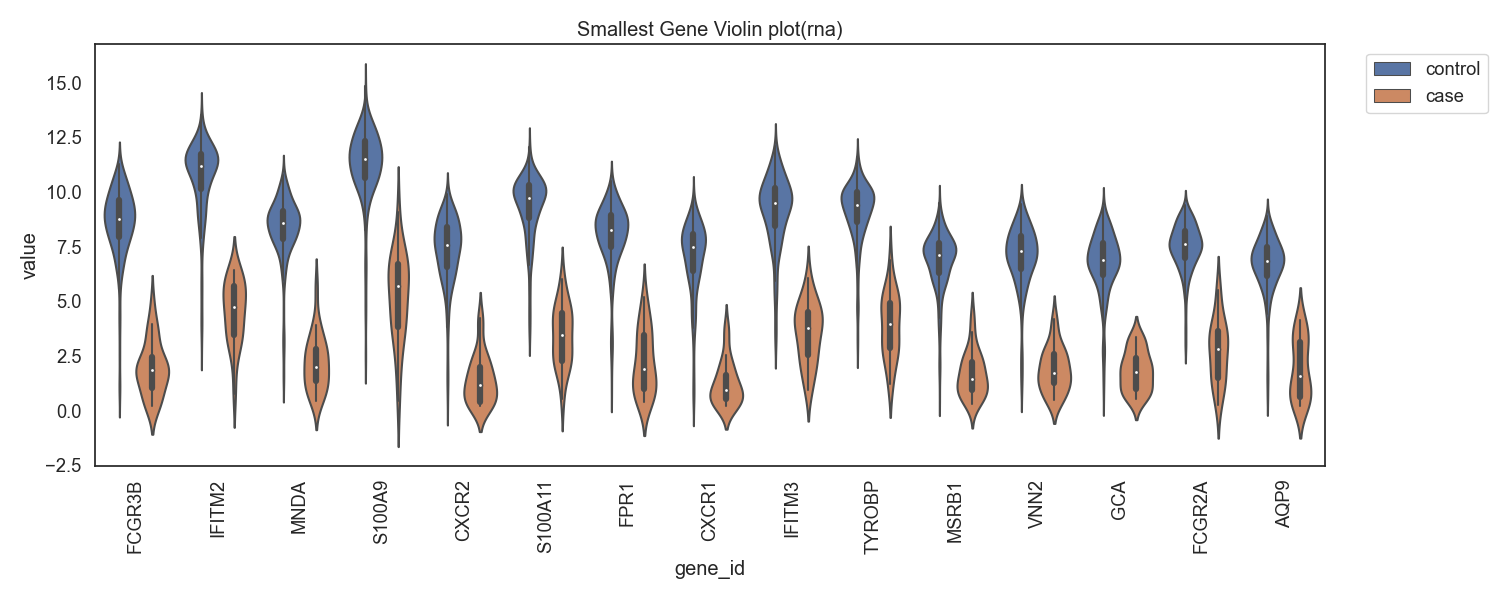
Fig14. RNA발현량의 차가 음으로 큰 gene 바이올린 플랏
DNA 메틸화와 유전자 발현과의 관계
Methyl-seq 분석 결과인 유전자별 DNA 메틸화 결과와 RNA-seq 분석 결과인 유전자별 RNA 발현 결과를 이용하여 어떠한 상관관계가 있는지 확인합니다. 유전자별 상관계수를 구하고, Case/control 그룹간 분포를 확인합니다.
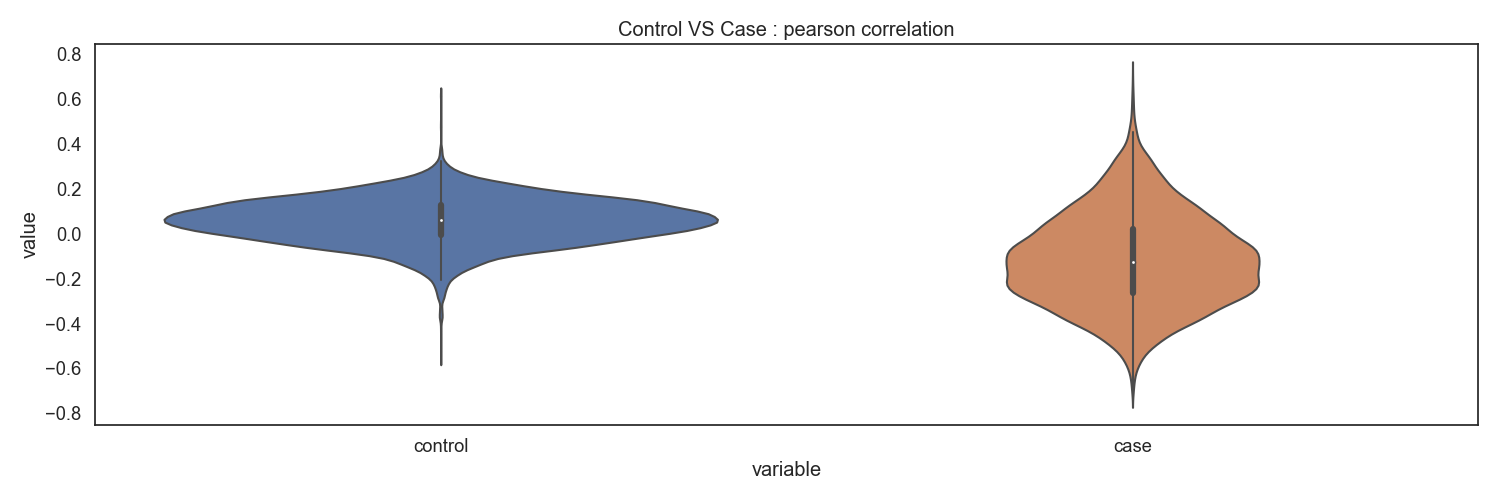
Fig15. Control vs Case : pearson correlation
DNA 메틸화와 RNA 발현량은 음의 상관관계가 있다고 알려져 있습니다. 음의 상관관계가 높은 상위 8개 유전자들을 확인합니다.
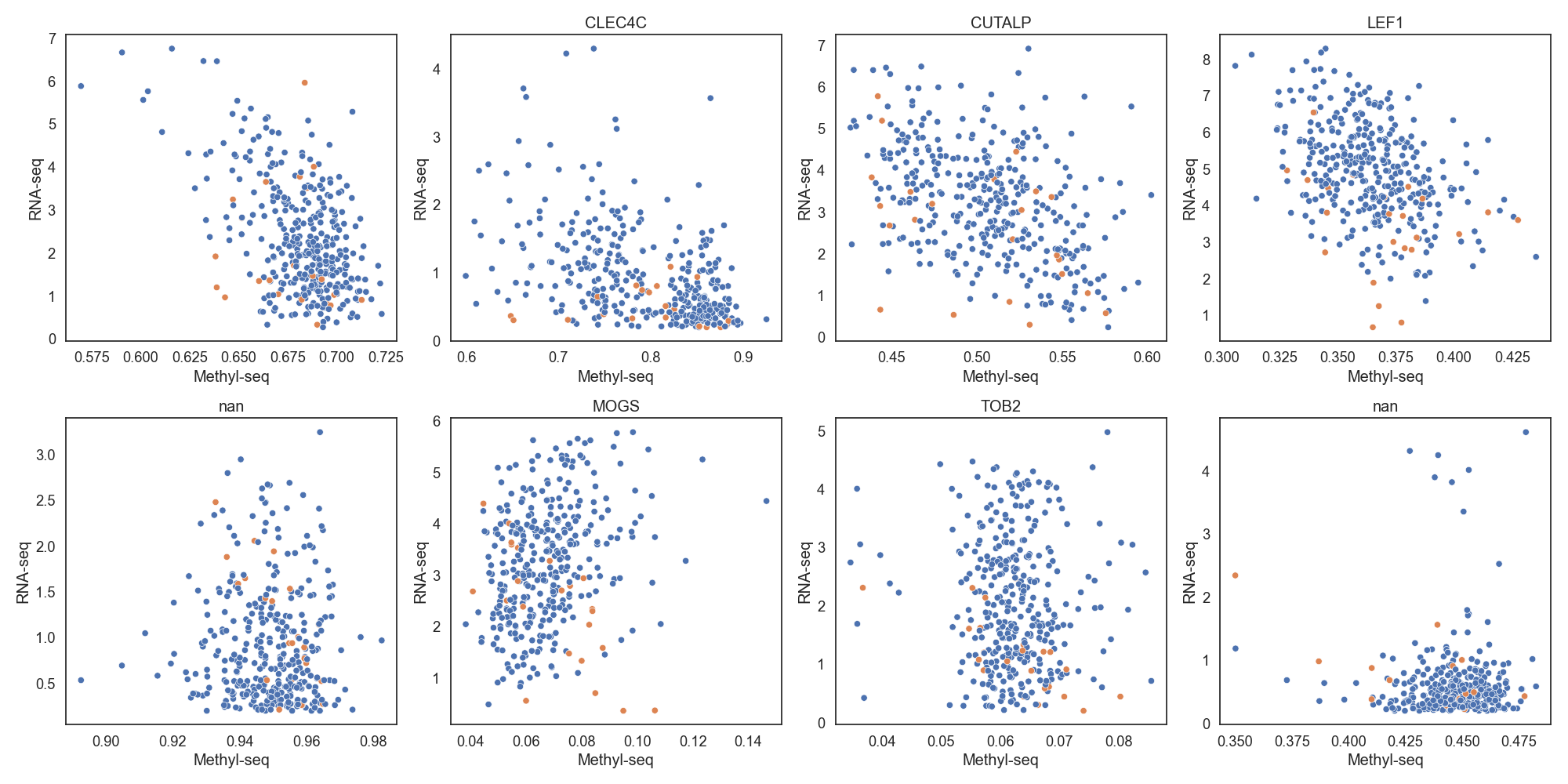
Fig16. RNA_seq, Methyl_seq scatter plot
Microbiome 분석
Case/control 각각 마이크로바이옴 풍부도 분포가 어떻게 차이나는지 확인합니다. 또한 이를 Phylum 별로 구분하여, 어떠한 미생물 계통이 더 많은지 확인합니다.
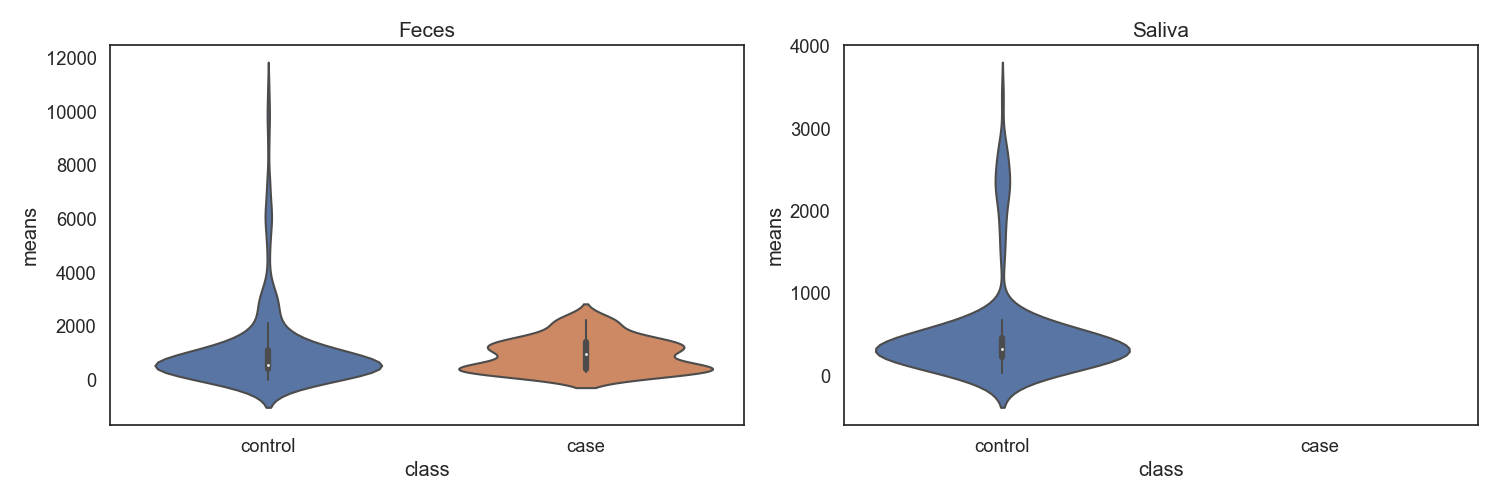
Fig17. Case vs. Control 전체 풍부도 특징
Case/control 각각 마이크로바이옴 다양성 차이가 어떠한지 확인합니다.
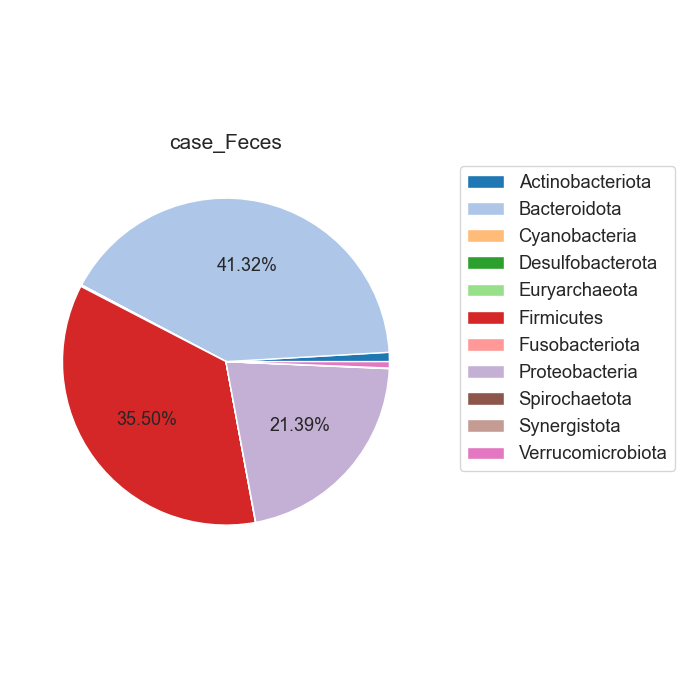
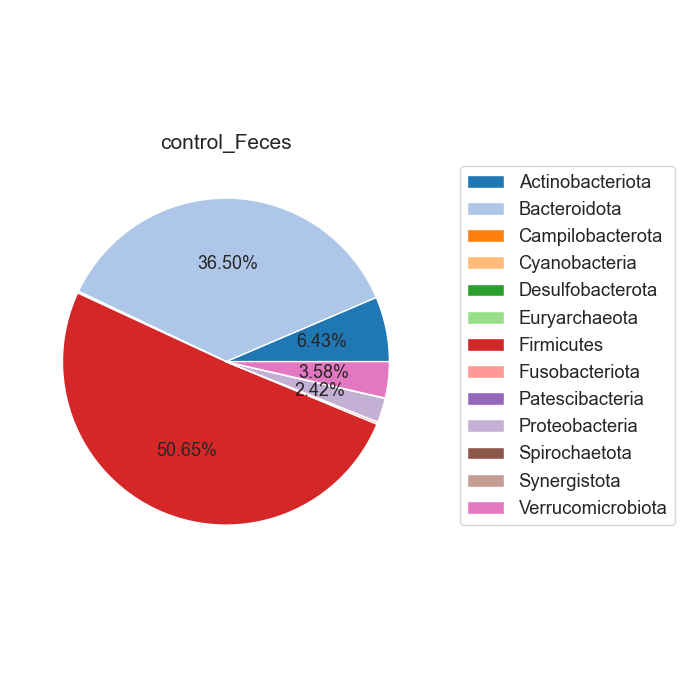
Fig18. Phylum 별 Microbiome 풍부도 특징
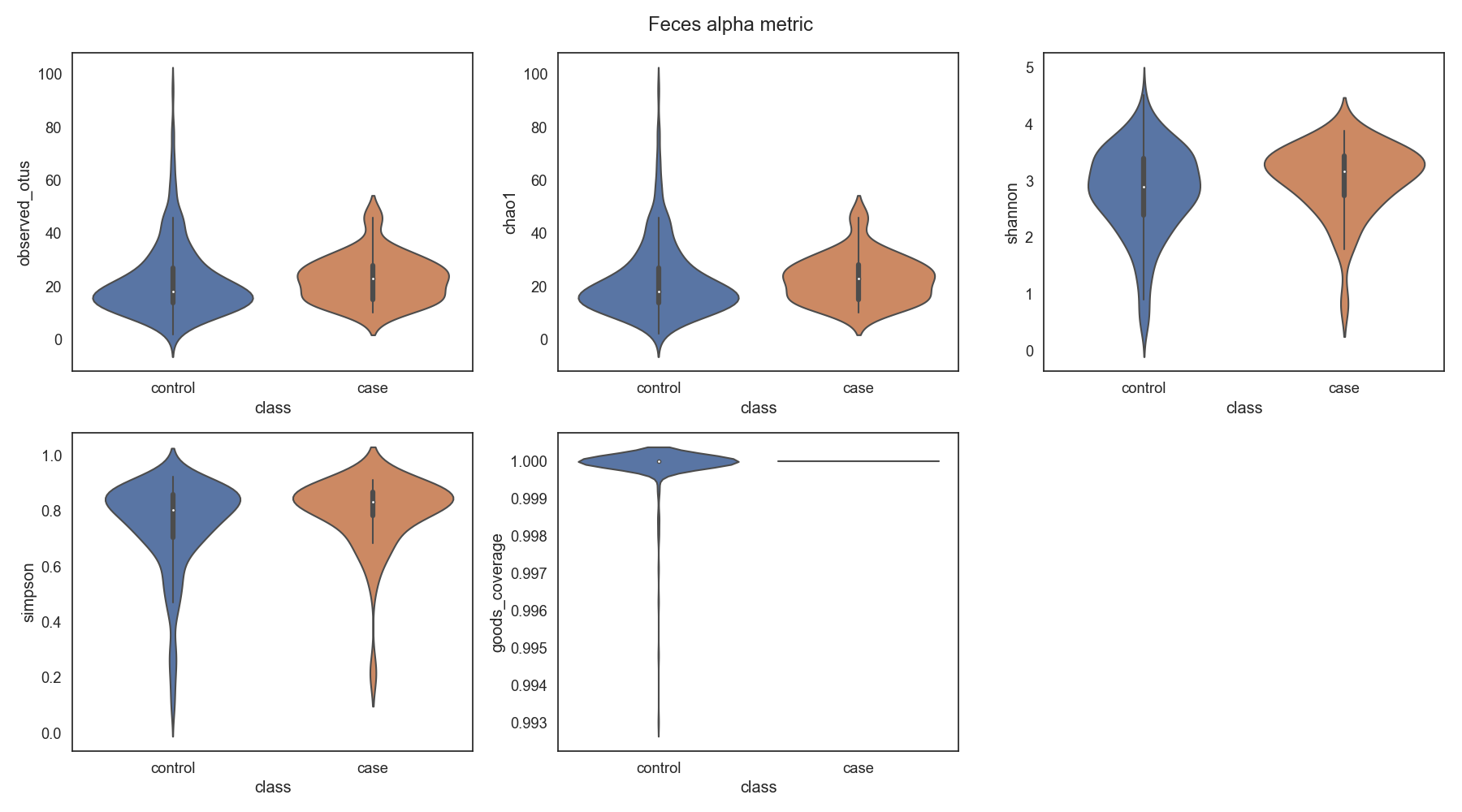
Fig19. Microbiome 다양성 분석
대장암 그룹 확인
헬빅쇼 데이터셋의 대장암 그룹은 "CCC" 입니다. 전체 환자수에서 위암이 차지하는 비율을 파이차트로 확인합니다. 그리고, case 그룹을 위암그룹으로 하고 나머지는 control 로 하여, 비교 분석을 수행합니다.
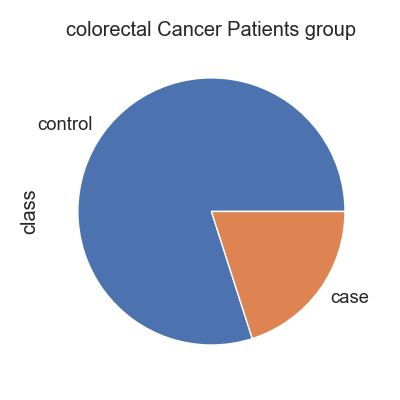
Fig1. 환자 그룹별 파이차트
변이 시그니처 분석
WGS 데이터로 부터, 변이 종류별 변이 갯수를 카운팅한 후, 이를 NMF 분석하여 7개의 시그니처를 생성합니다. case, control 각각 시그니처 분포를 확인합니다.
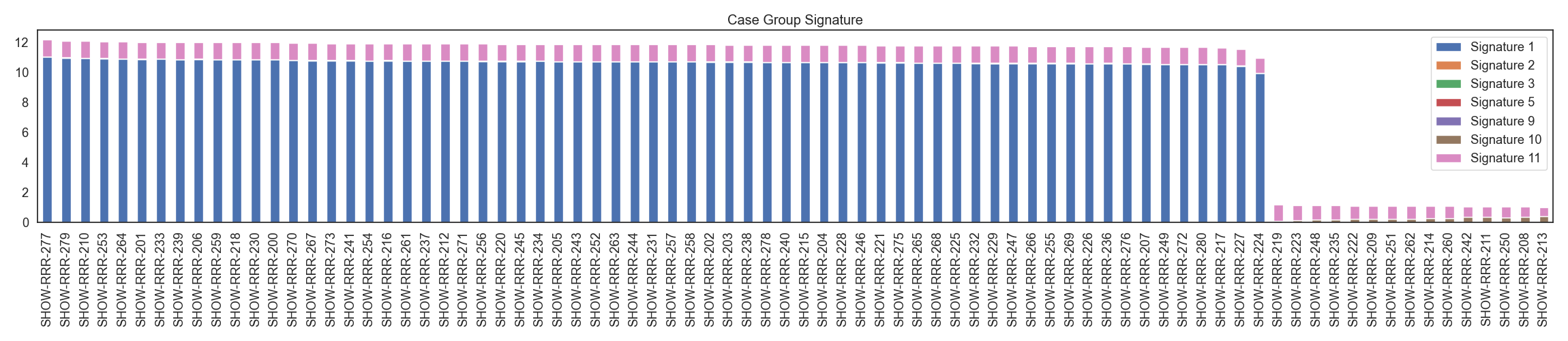
Fig2. Case 그룹 시그니처 분포
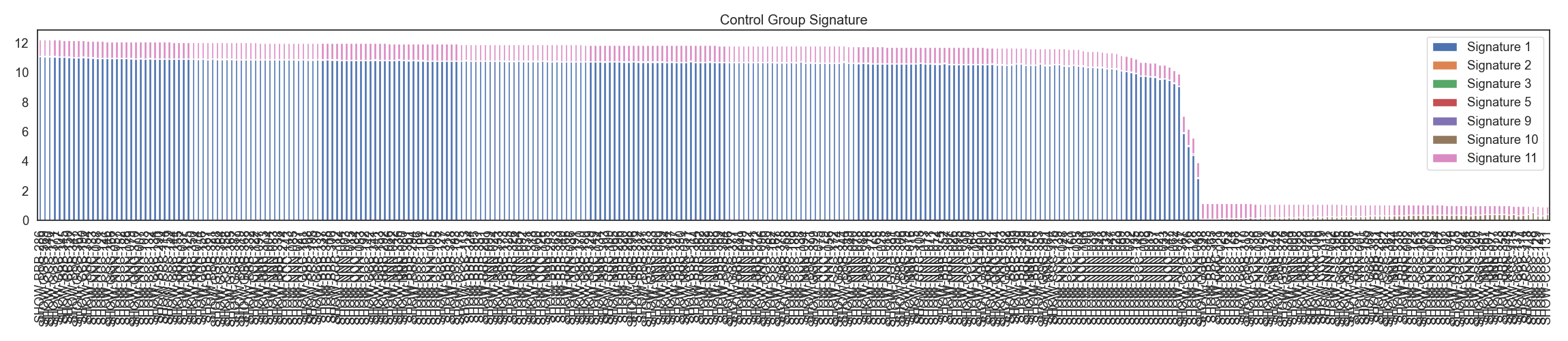
Fig3. Control 그룹 시그니처 분포
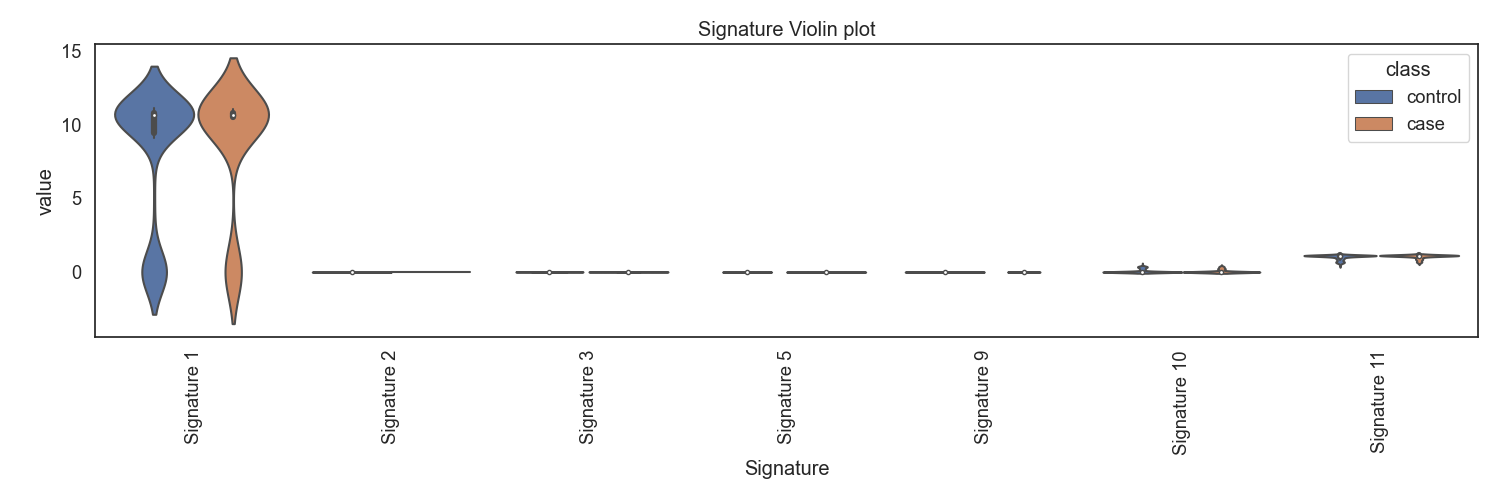
Fig4. 시그니처별 바이올린 플랏
DNA 메틸화 비율
Methyl-seq 데이터로 부터, 유전자별 DNA 메틸화 비율을 계산하고, 환자별로 평균을 계산한 후, 비교합니다.
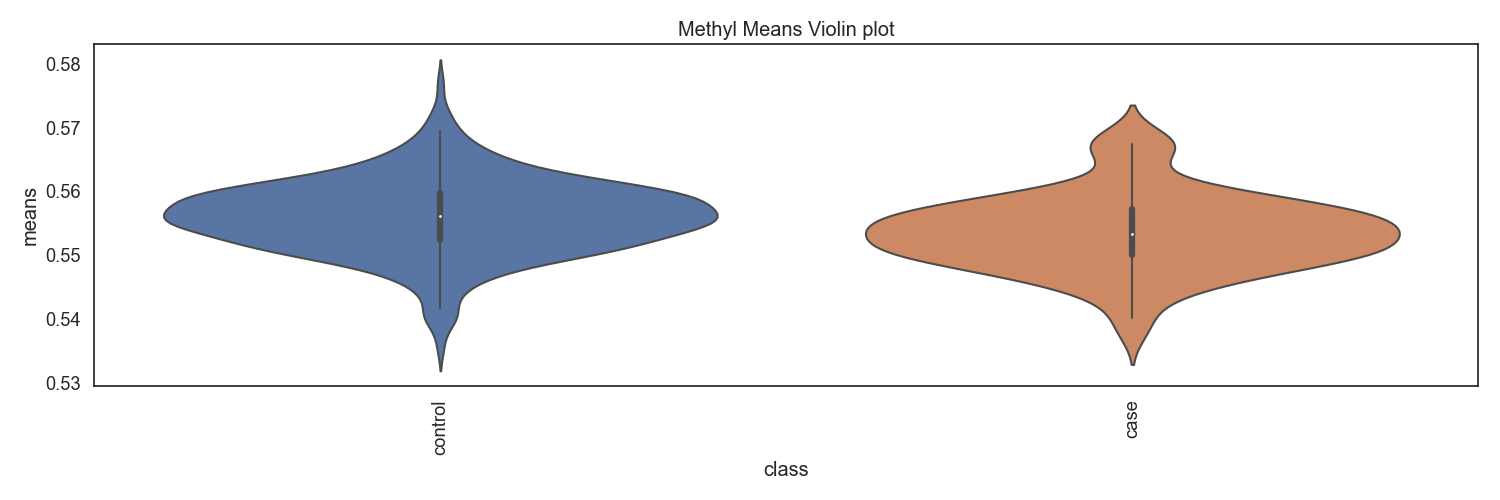
Fig5. 클래스별 메틸화 바이올린 플랏
Case/control 그룹간 차이가 큰 유전자 상위 10개의 유전자목록은 다음과 같습니다.
Fig6. 메틸화의 차가 양으로 큰 gene
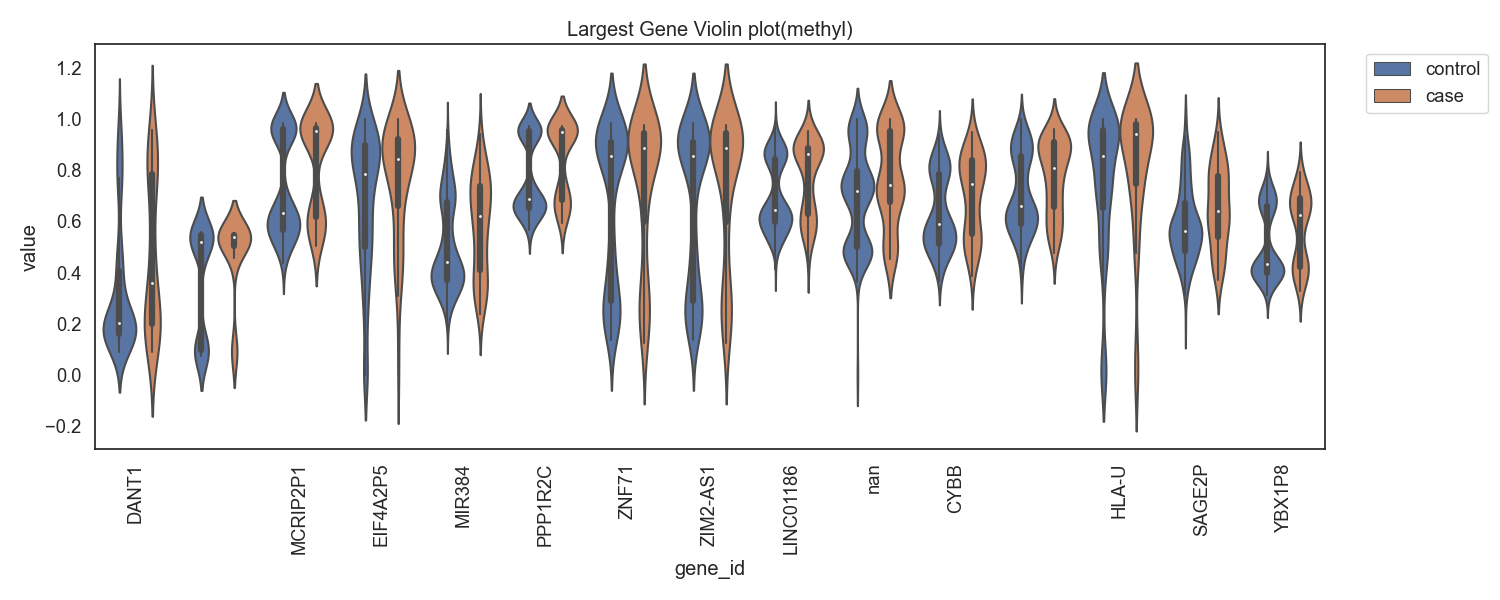
Fig7. 메틸화의 차가 양으로 큰 gene 바이올린 플랏
Fig8. 메틸화의 차가 음으로 큰 gene
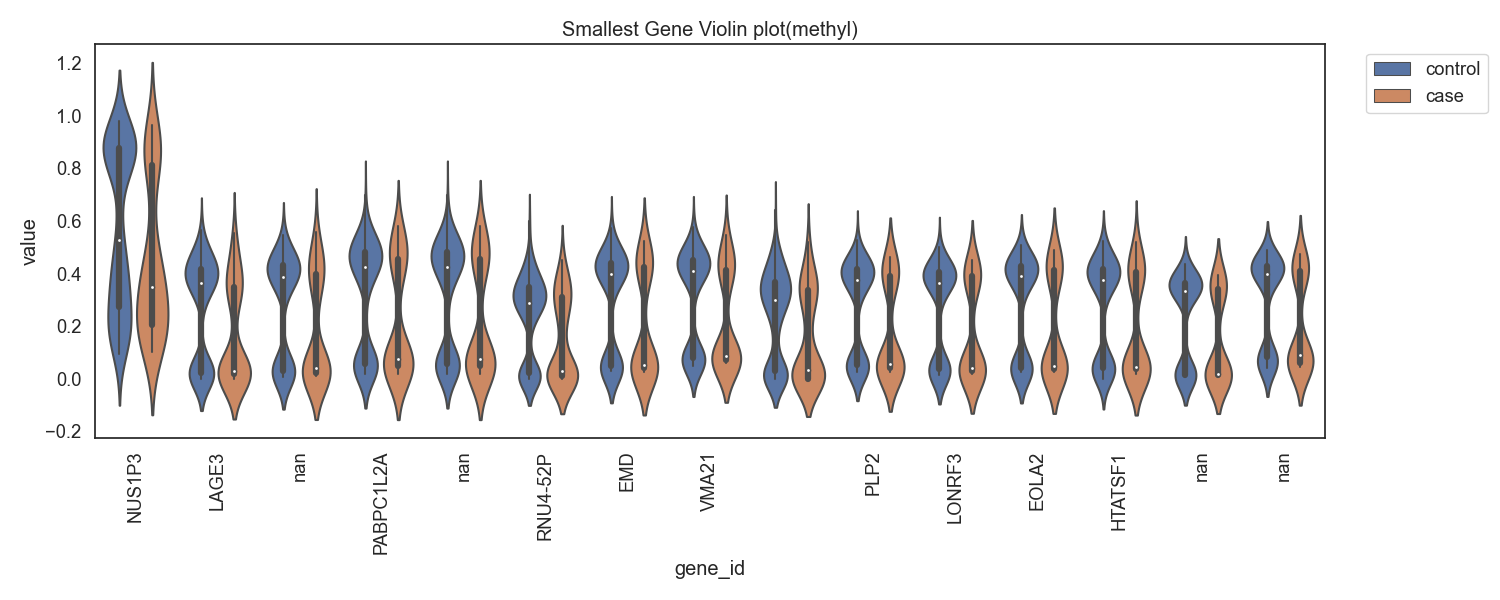
Fig9. 메틸화의 차가 음으로 큰 gene 바이올린 플랏
유전자 발현 분석
RNA-seq 분석을 통해 확인한 TPM 자료로 부터, Case/control 그룹간 전체 환자별 유전자 발현 평균 분포를 확인합니다.
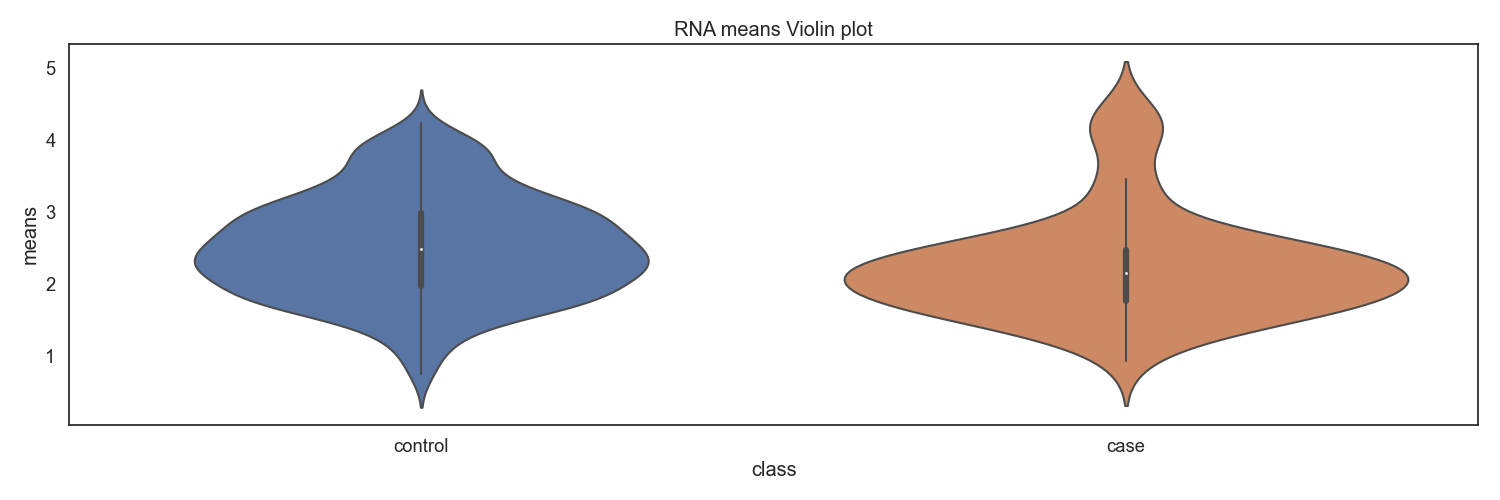
Fig10. 클래스별 RNA발현량 바이올린 플랏
Case/control 그룹간 유전자 발현 차이가 높은 유전자들의 목록을 확인합니다.
Fig11. RNA발현량의 차가 음으로 큰 gene
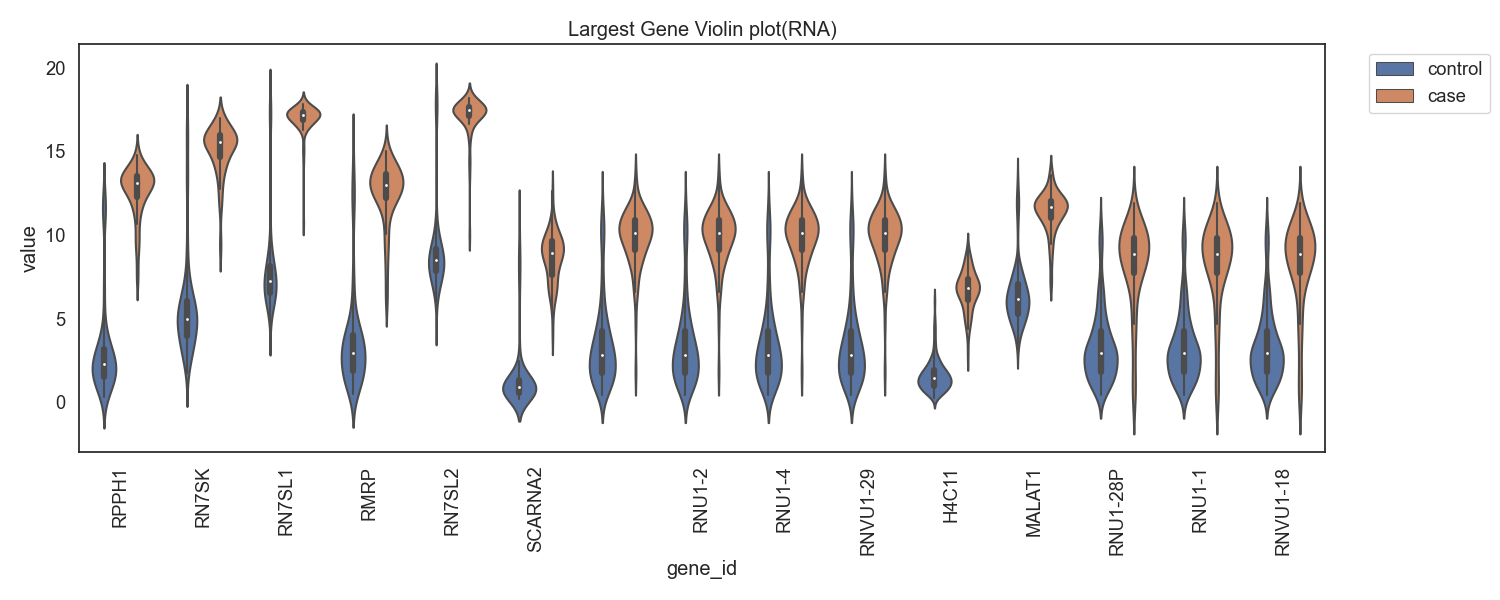
Fig12. RNA발현량의 차가 양으로 큰 gene 바이올린 플랏
Fig13. RNA발현량의 차가 음으로 큰 gene
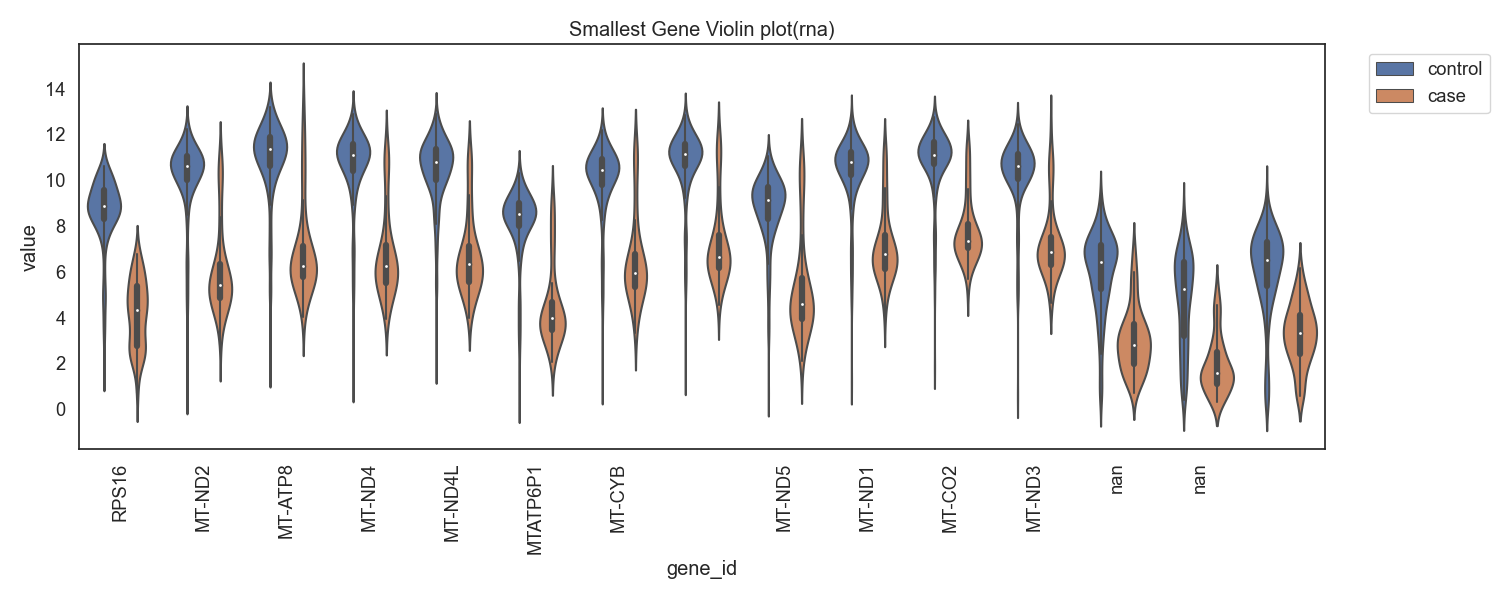
Fig14. RNA발현량의 차가 음으로 큰 gene 바이올린 플랏
DNA 메틸화와 유전자 발현과의 관계
Methyl-seq 분석 결과인 유전자별 DNA 메틸화 결과와 RNA-seq 분석 결과인 유전자별 RNA 발현 결과를 이용하여 어떠한 상관관계가 있는지 확인합니다. 유전자별 상관계수를 구하고, Case/control 그룹간 분포를 확인합니다.
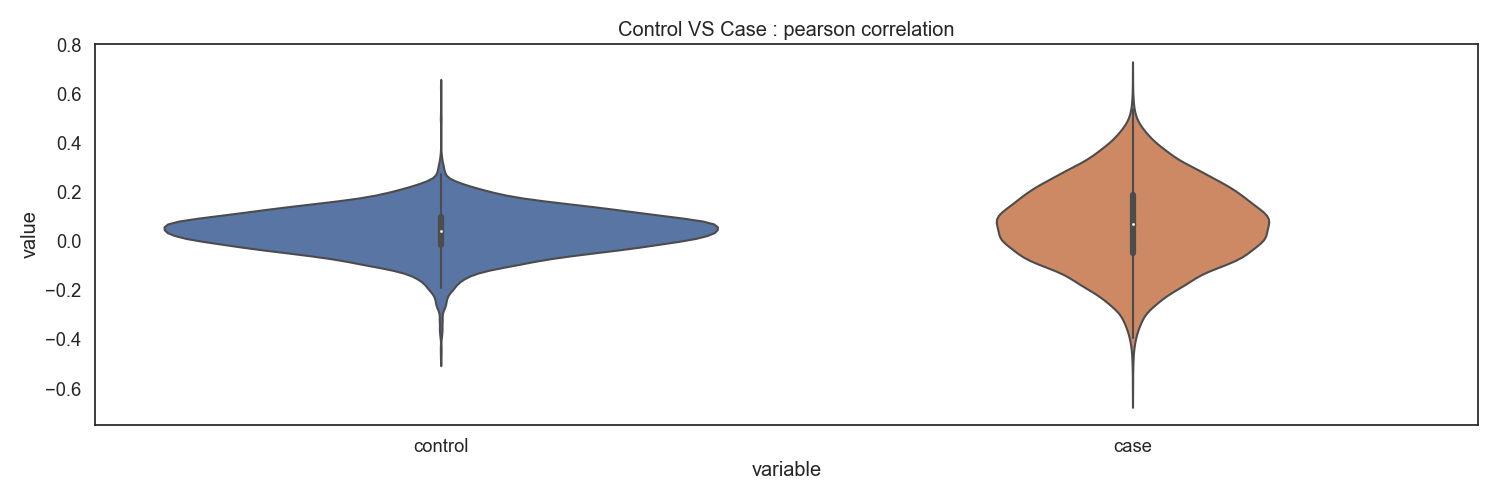
Fig15. Control vs Case : pearson correlation
DNA 메틸화와 RNA 발현량은 음의 상관관계가 있다고 알려져 있습니다. 음의 상관관계가 높은 상위 8개 유전자들을 확인합니다.
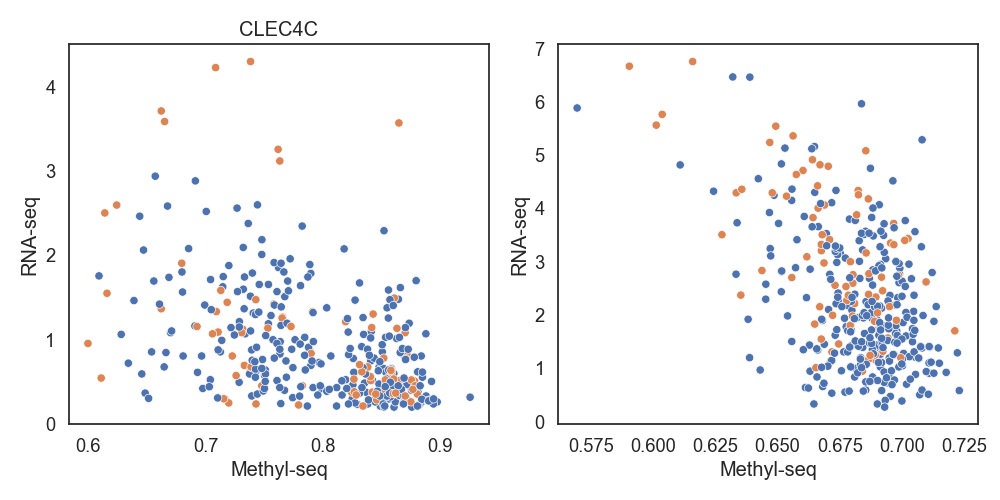
Fig16. RNA_seq, Methyl_seq scatter plot
Microbiome 분석
Case/control 각각 마이크로바이옴 풍부도 분포가 어떻게 차이나는지 확인합니다. 또한 이를 Phylum 별로 구분하여, 어떠한 미생물 계통이 더 많은지 확인합니다.
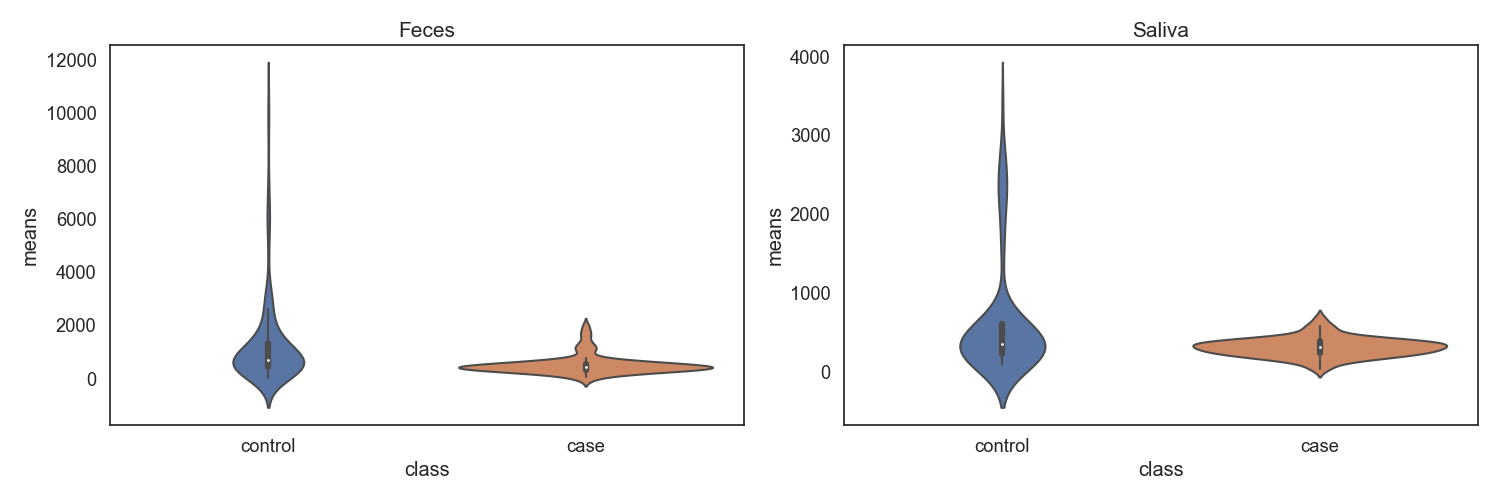
Fig17. Case vs. Control 전체 풍부도 특징
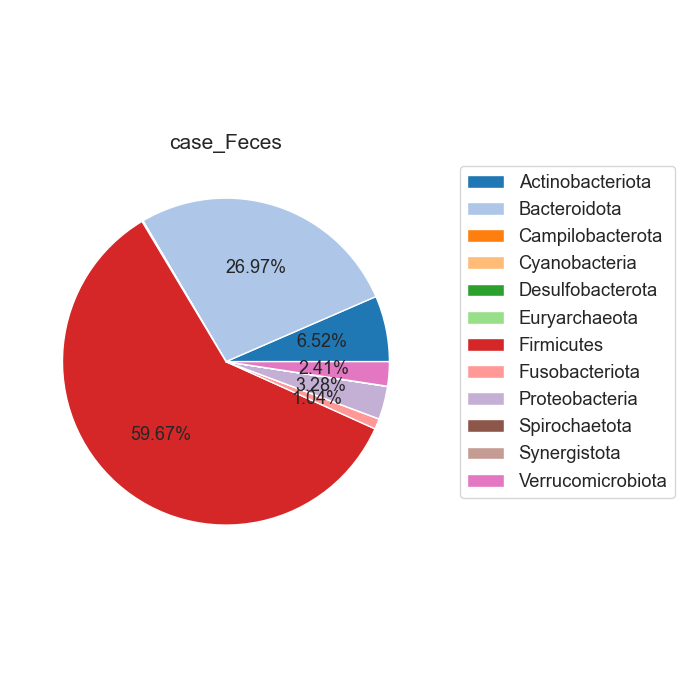
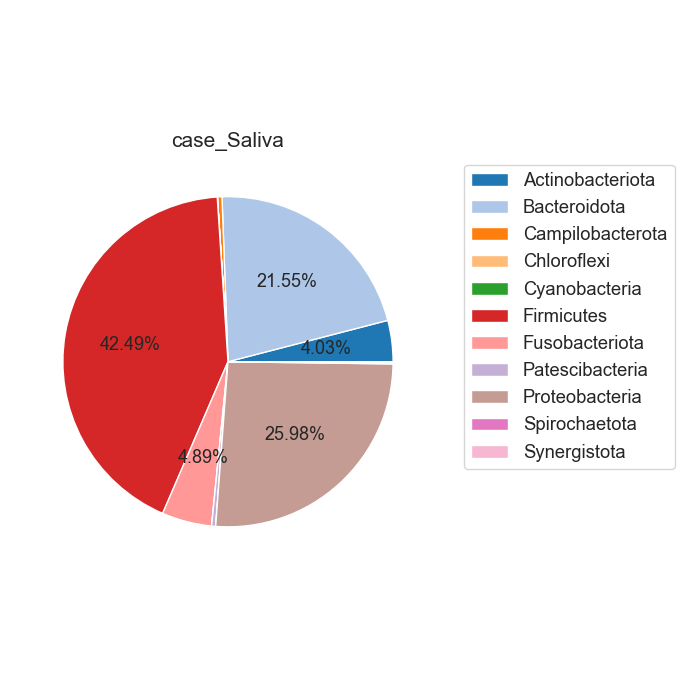
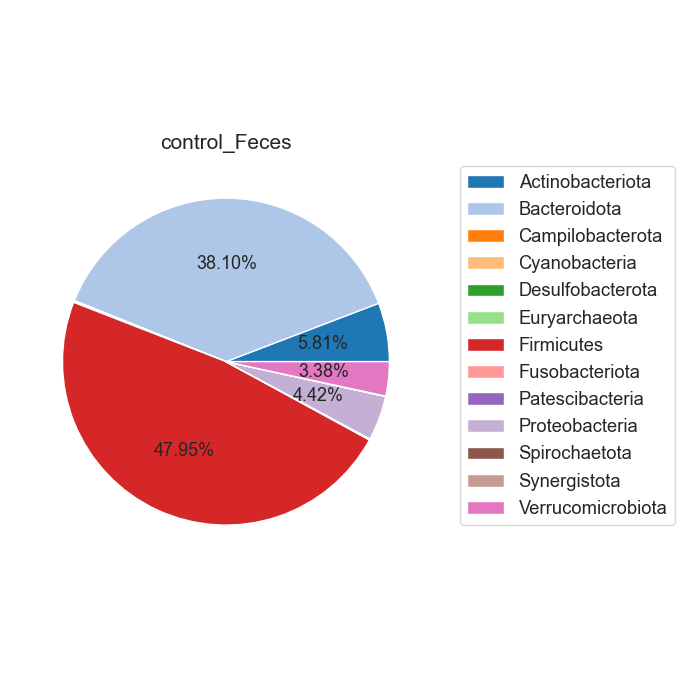
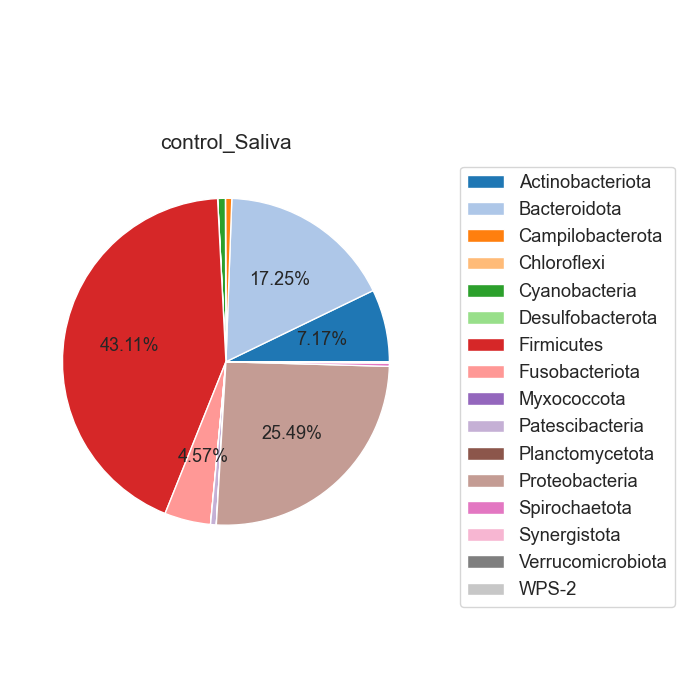
Fig18. Phylum 별 Microbiome 풍부도 특징
Case/control 각각 마이크로바이옴 다양성 차이가 어떠한지 확인합니다.
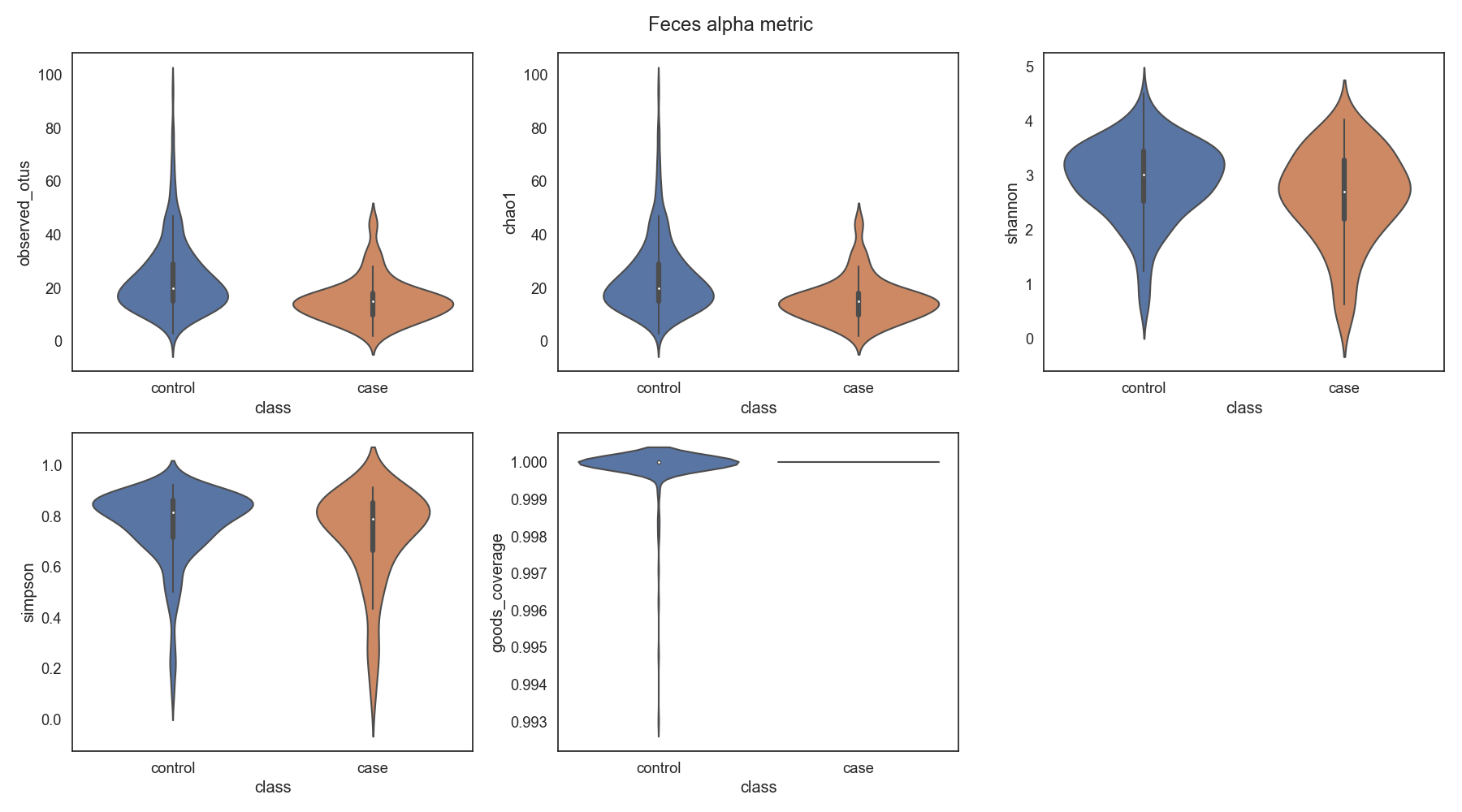
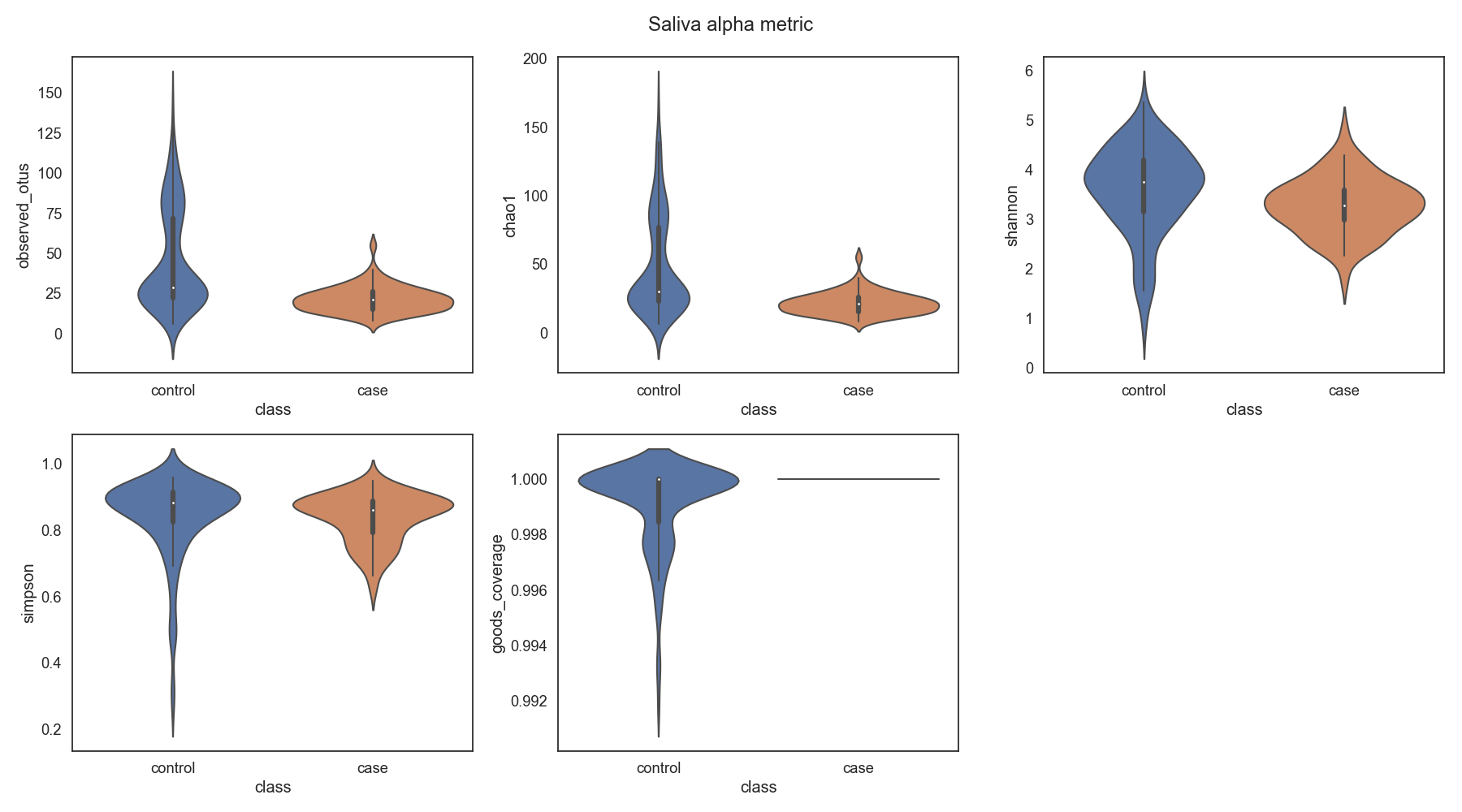
Fig19. Microbiome 다양성 분석
유방암 그룹 확인
헬빅쇼 데이터셋의 위암 그룹은 "BBB" 입니다. 전체 환자수에서 위암이 차지하는 비율을 파이차트로 확인합니다. 그리고, case 그룹을 위암그룹으로 하고 나머지는 control 로 하여, 비교 분석을 수행합니다.
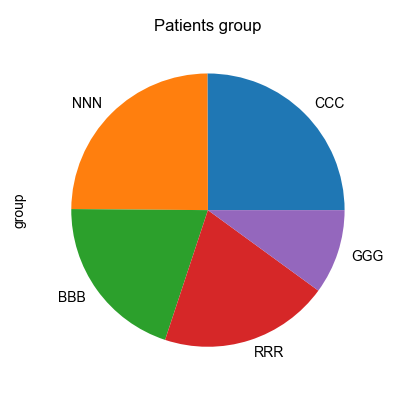
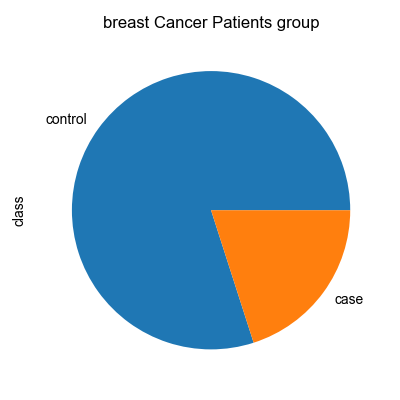
Fig1. 환자 그룹별 파이차트
변이 시그니처 분석
WGS 데이터로 부터, 변이 종류별 변이 갯수를 카운팅한 후, 이를 NMF 분석하여 7개의 시그니처를 생성합니다. case, control 각각 시그니처 분포를 확인합니다.
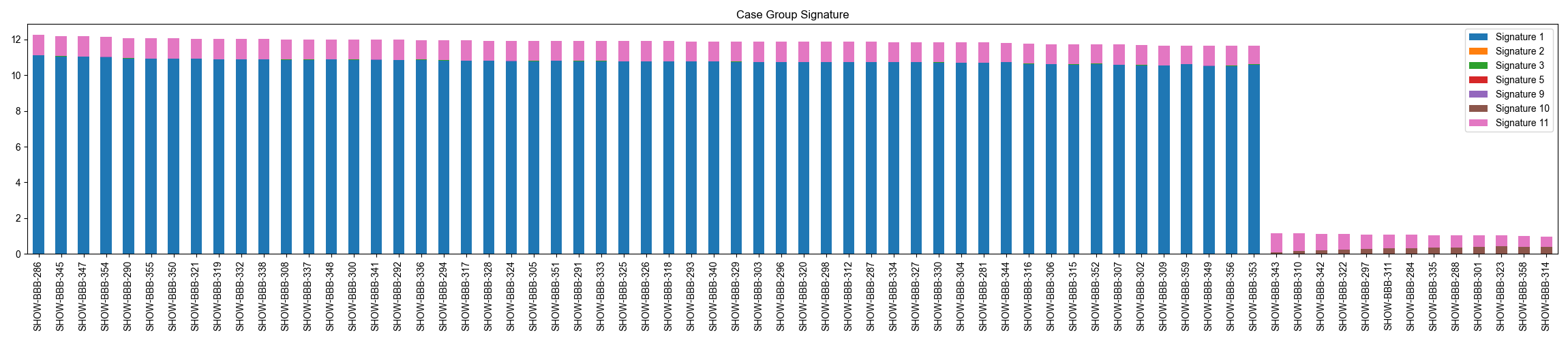
Fig2. Case 그룹 시그니처 분포
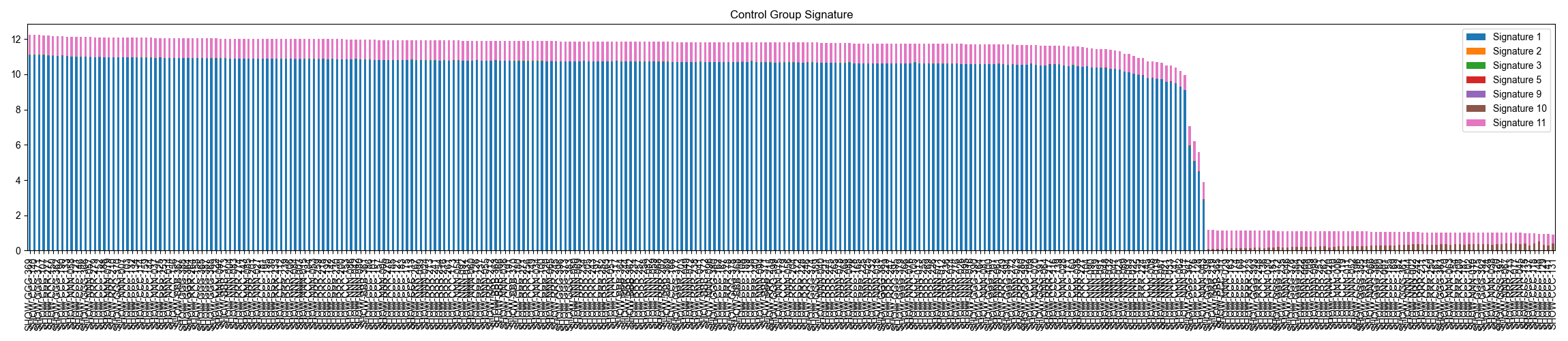
Fig3. Control 그룹 시그니처 분포
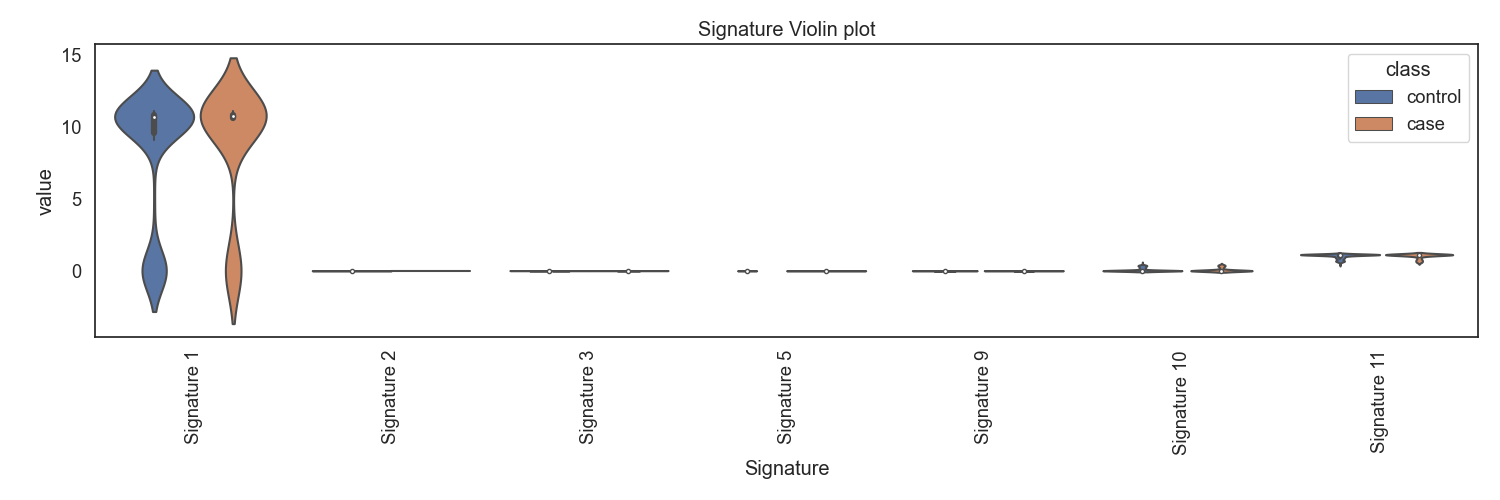
Fig4. 시그니처별 바이올린 플랏
DNA 메틸화 비율
Methyl-seq 데이터로 부터, 유전자별 DNA 메틸화 비율을 계산하고, 환자별로 평균을 계산한 후, 비교합니다.
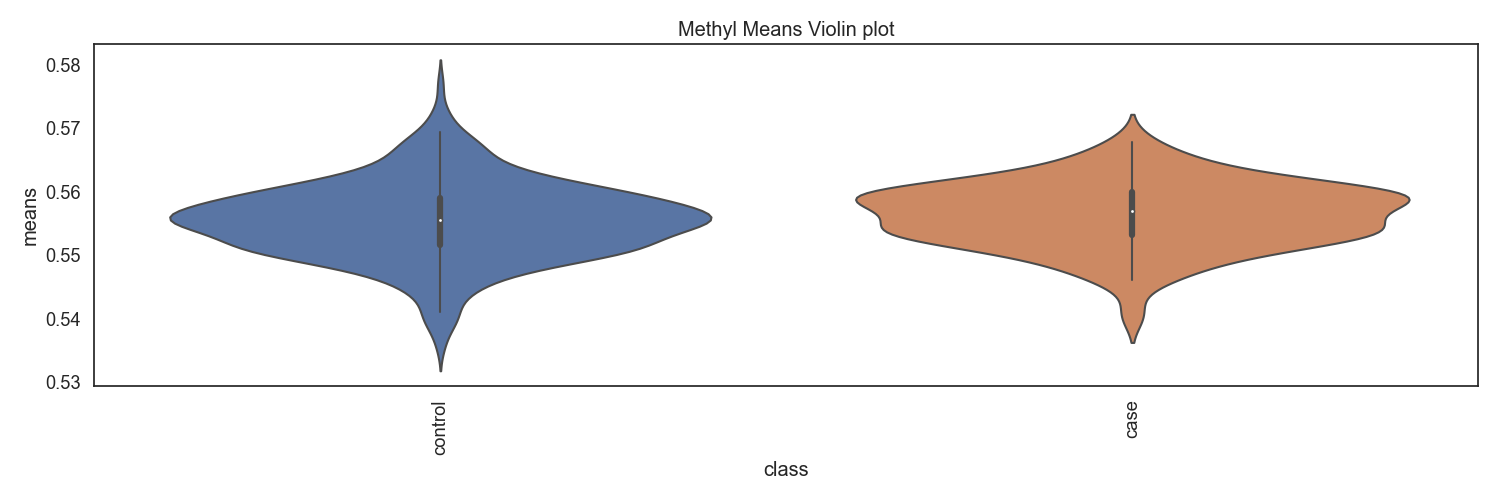
Fig5. 클래스별 메틸화 바이올린 플랏
Case/control 그룹간 차이가 큰 유전자 상위 10개의 유전자목록은 다음과 같습니다.
Fig6. 메틸화의 차가 양으로 큰 gene
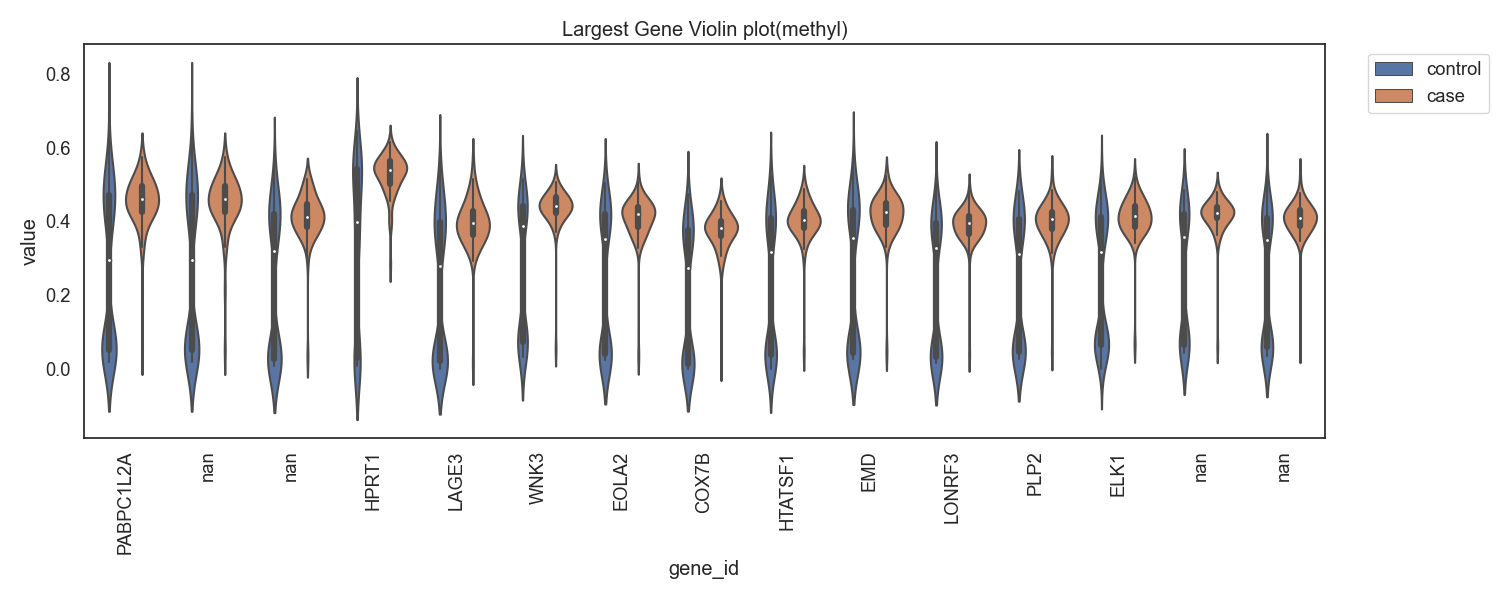
Fig7. 메틸화의 차가 양으로 큰 gene 바이올린 플랏
Fig8. 메틸화의 차가 음으로 큰 gene
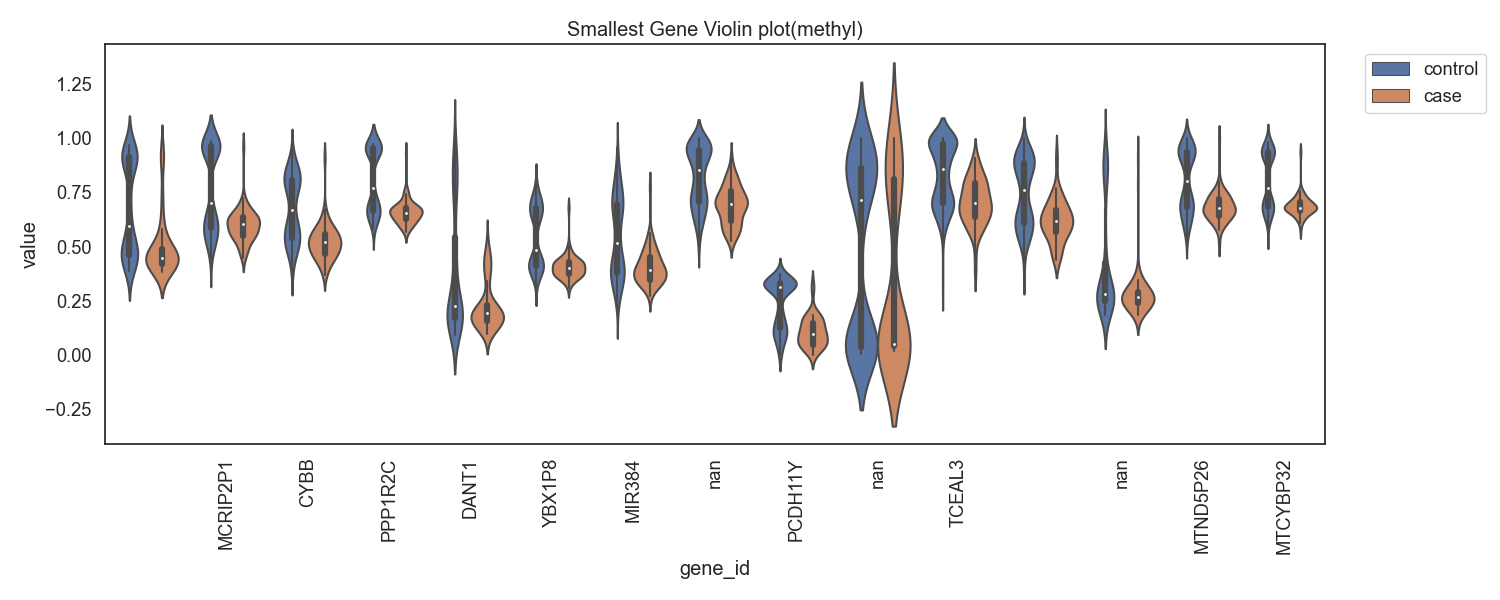
Fig9. 메틸화의 차가 음으로 큰 gene 바이올린 플랏
유전자 발현 분석
RNA-seq 분석을 통해 확인한 TPM 자료로 부터, Case/control 그룹간 전체 환자별 유전자 발현 평균 분포를 확인합니다.
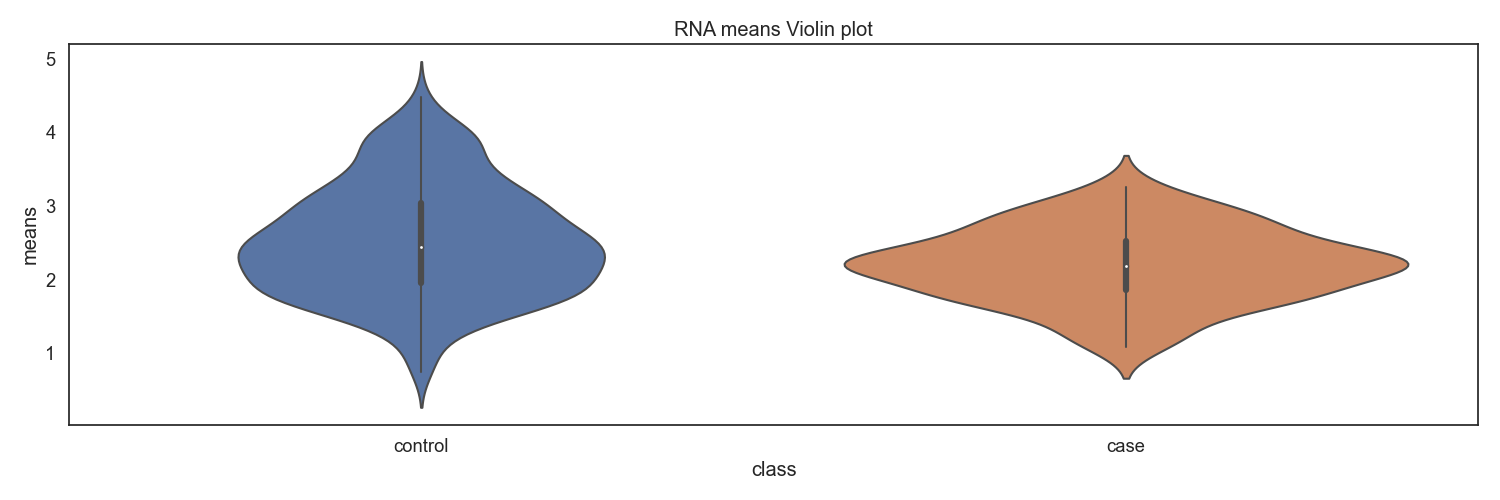
Fig10. 클래스별 RNA발현량 바이올린 플랏
Case/control 그룹간 유전자 발현 차이가 높은 유전자들의 목록을 확인합니다.
Fig11. RNA발현량의 차가 음으로 큰 gene
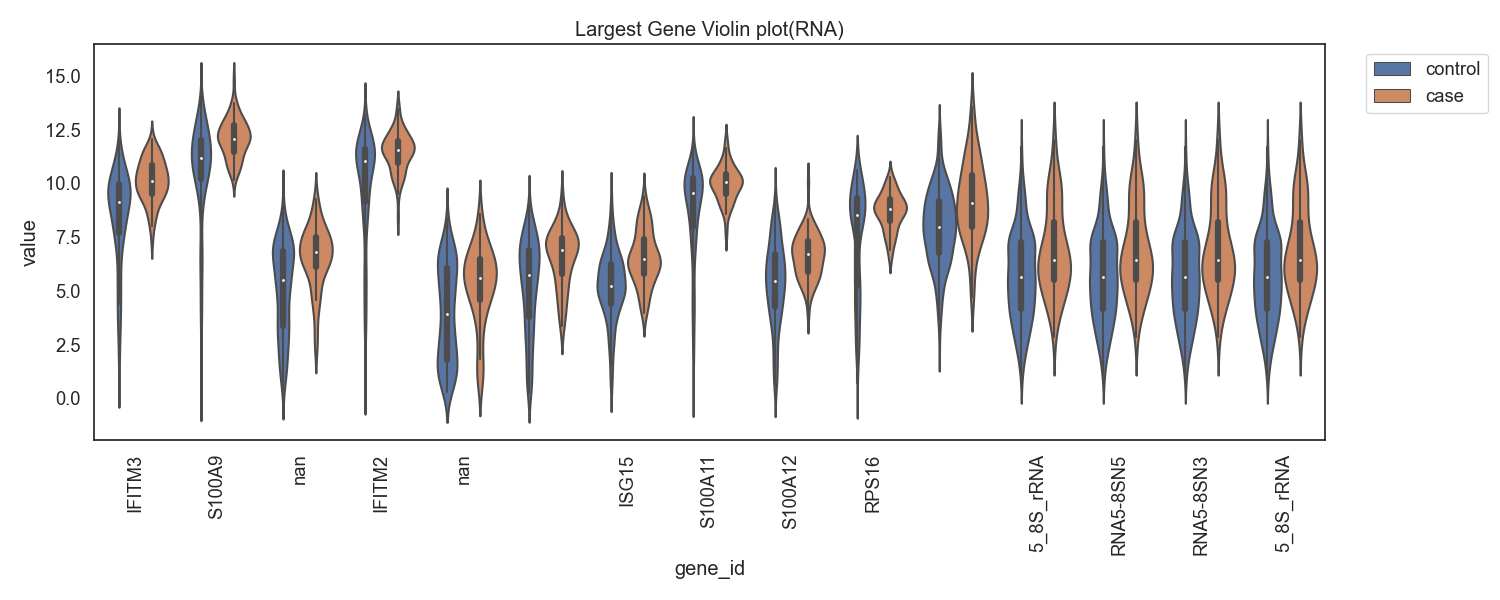
Fig12. RNA발현량의 차가 양으로 큰 gene 바이올린 플랏
Fig13. RNA발현량의 차가 음으로 큰 gene
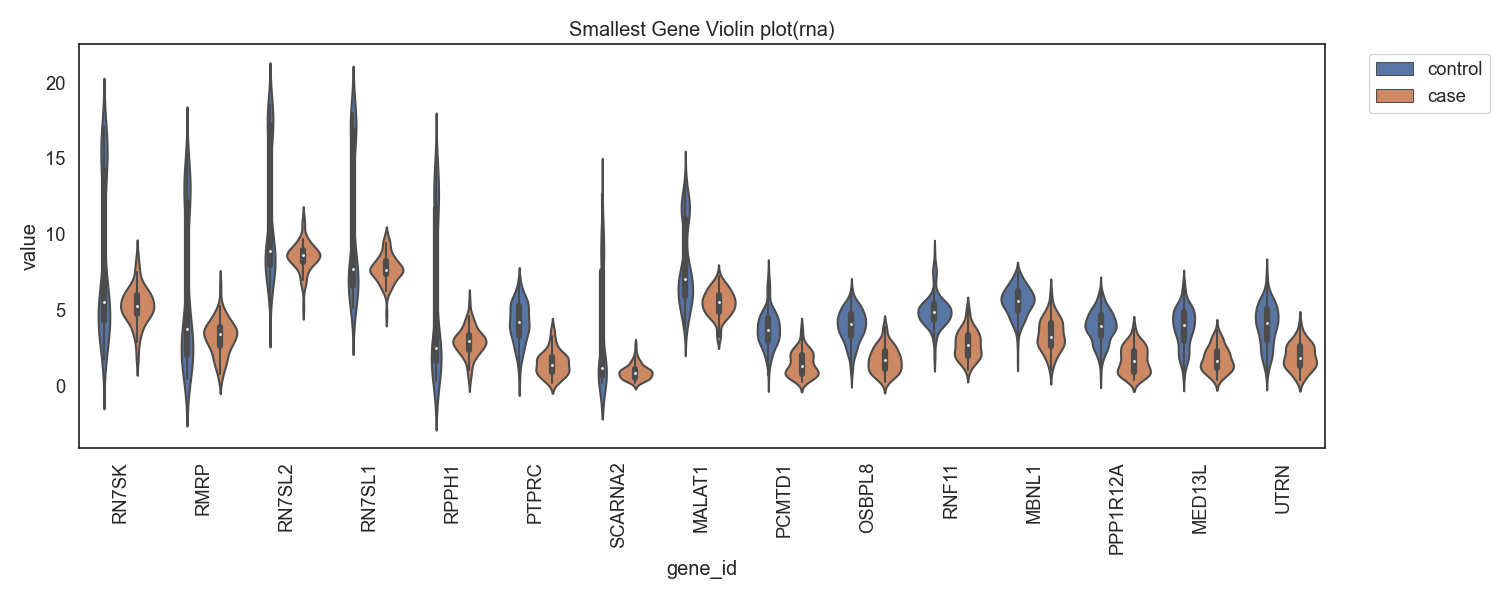
Fig14. RNA발현량의 차가 음으로 큰 gene 바이올린 플랏
DNA 메틸화와 유전자 발현과의 관계
Methyl-seq 분석 결과인 유전자별 DNA 메틸화 결과와 RNA-seq 분석 결과인 유전자별 RNA 발현 결과를 이용하여 어떠한 상관관계가 있는지 확인합니다. 유전자별 상관계수를 구하고, Case/control 그룹간 분포를 확인합니다.
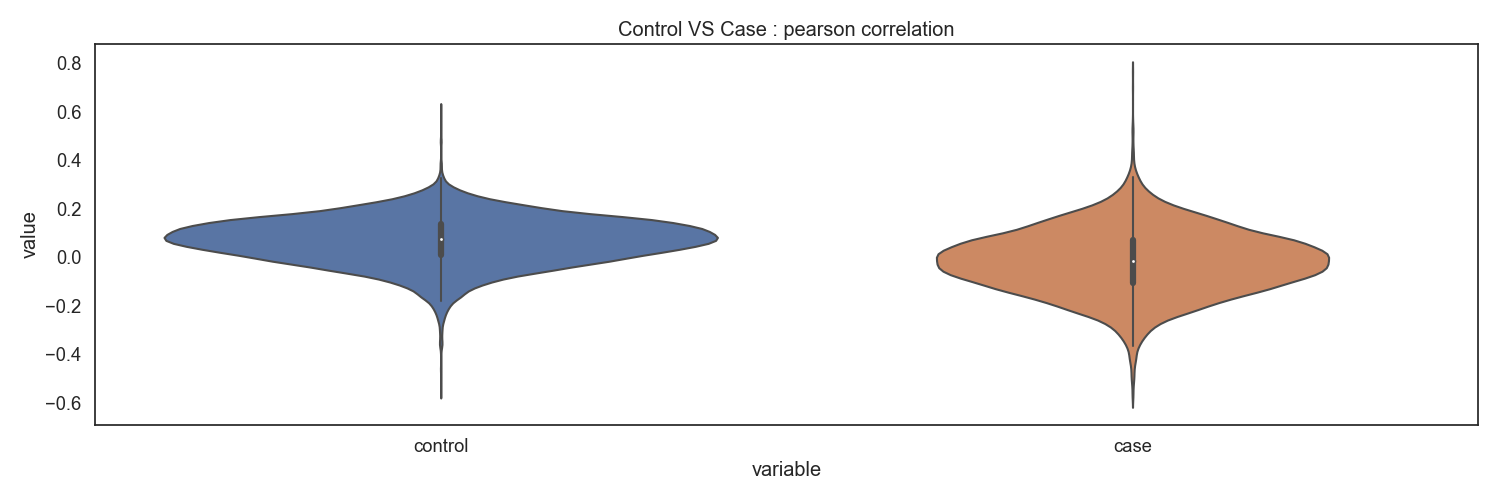
Fig15. Control vs Case : pearson correlation
DNA 메틸화와 RNA 발현량은 음의 상관관계가 있다고 알려져 있습니다. 음의 상관관계가 높은 상위 8개 유전자들을 확인합니다.
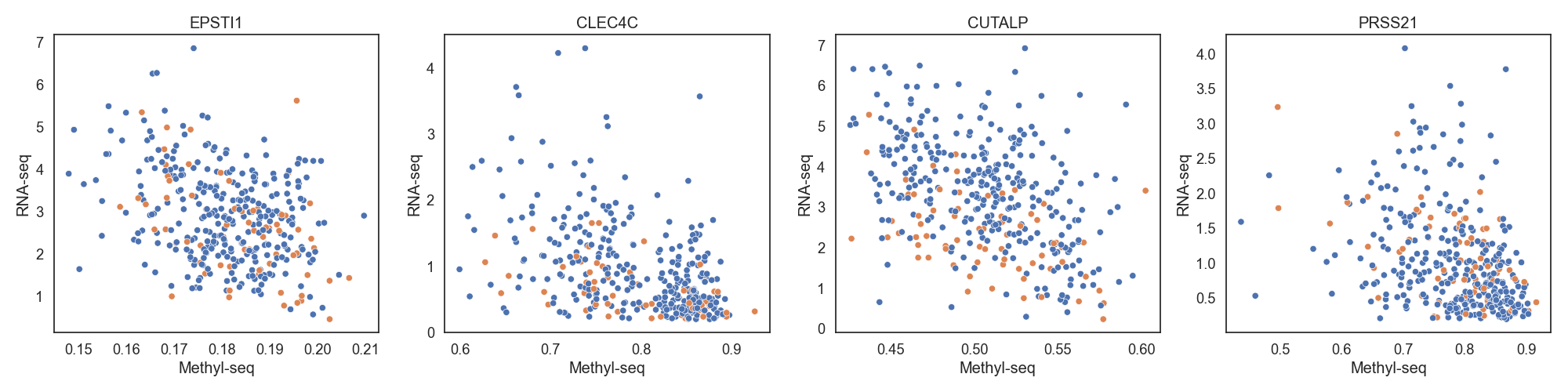
Fig16. RNA_seq, Methyl_seq scatter plot
Microbiome 분석
Case/control 각각 마이크로바이옴 풍부도 분포가 어떻게 차이나는지 확인합니다. 또한 이를 Phylum 별로 구분하여, 어떠한 미생물 계통이 더 많은지 확인합니다.
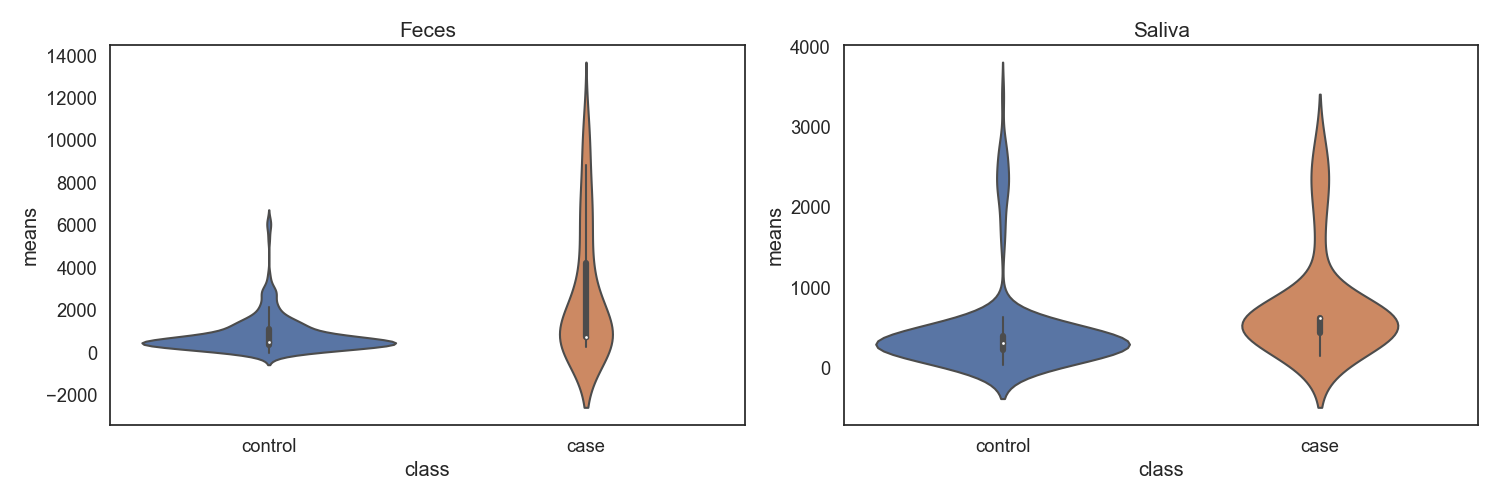
Fig17. Case vs. Control 전체 풍부도 특징
Case/control 각각 마이크로바이옴 다양성 차이가 어떠한지 확인합니다.
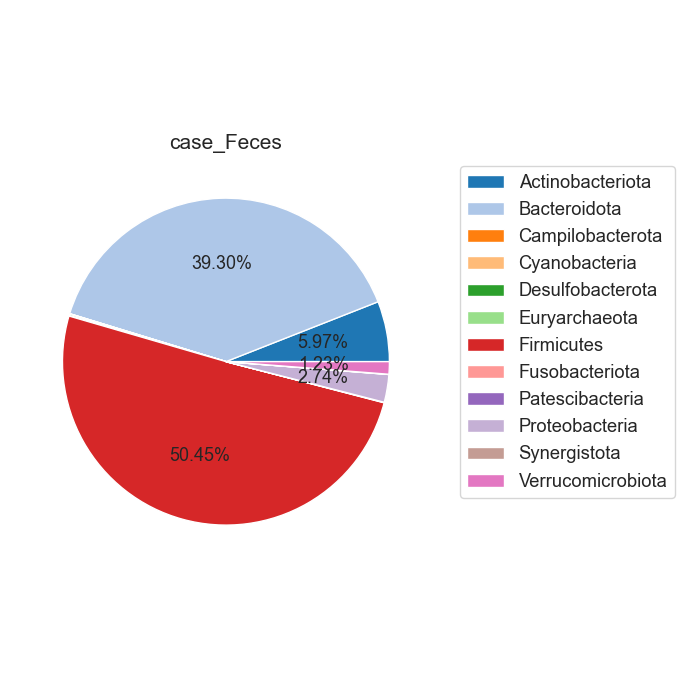
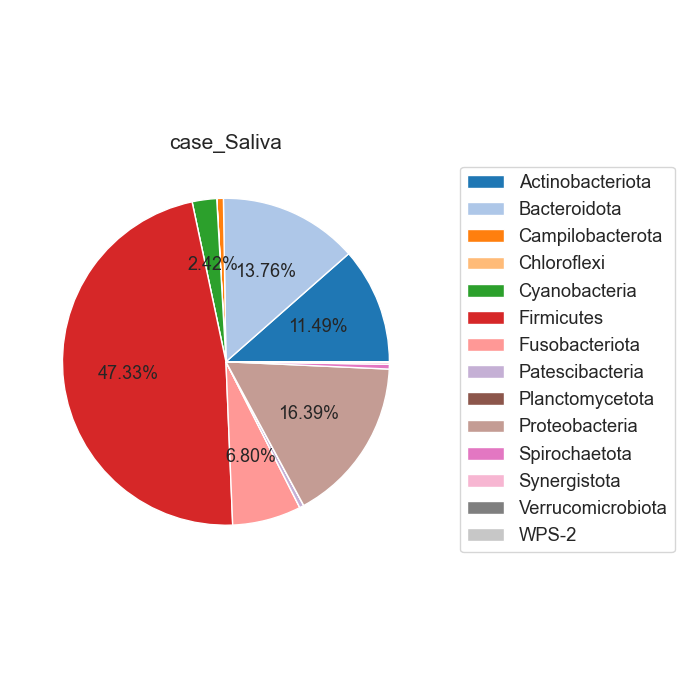
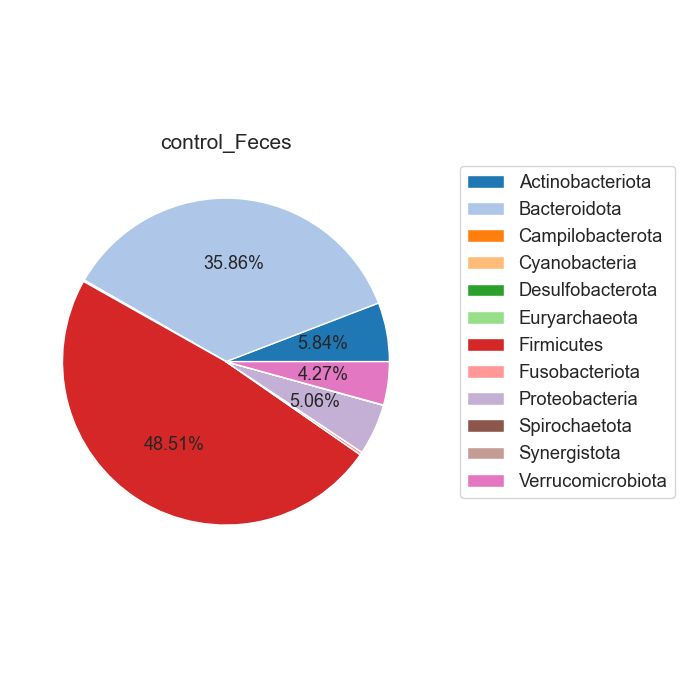
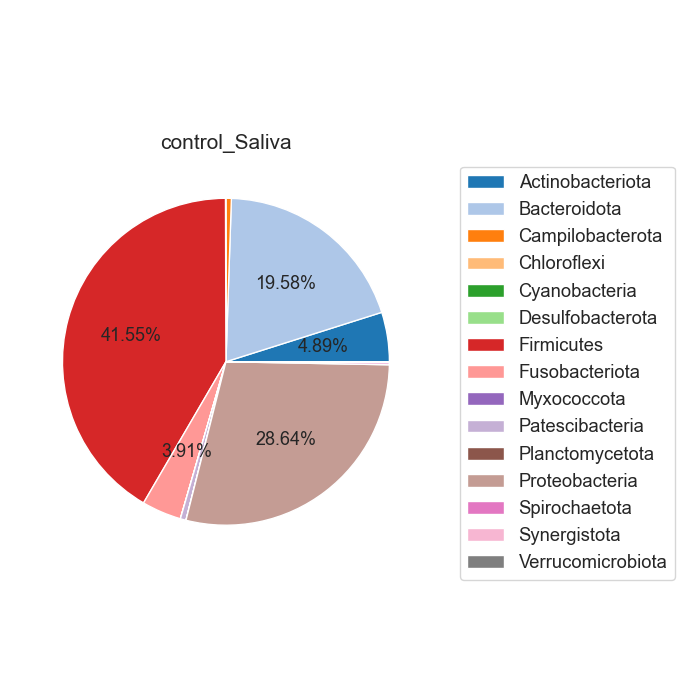
Fig18. Phylum 별 Microbiome 풍부도 특징
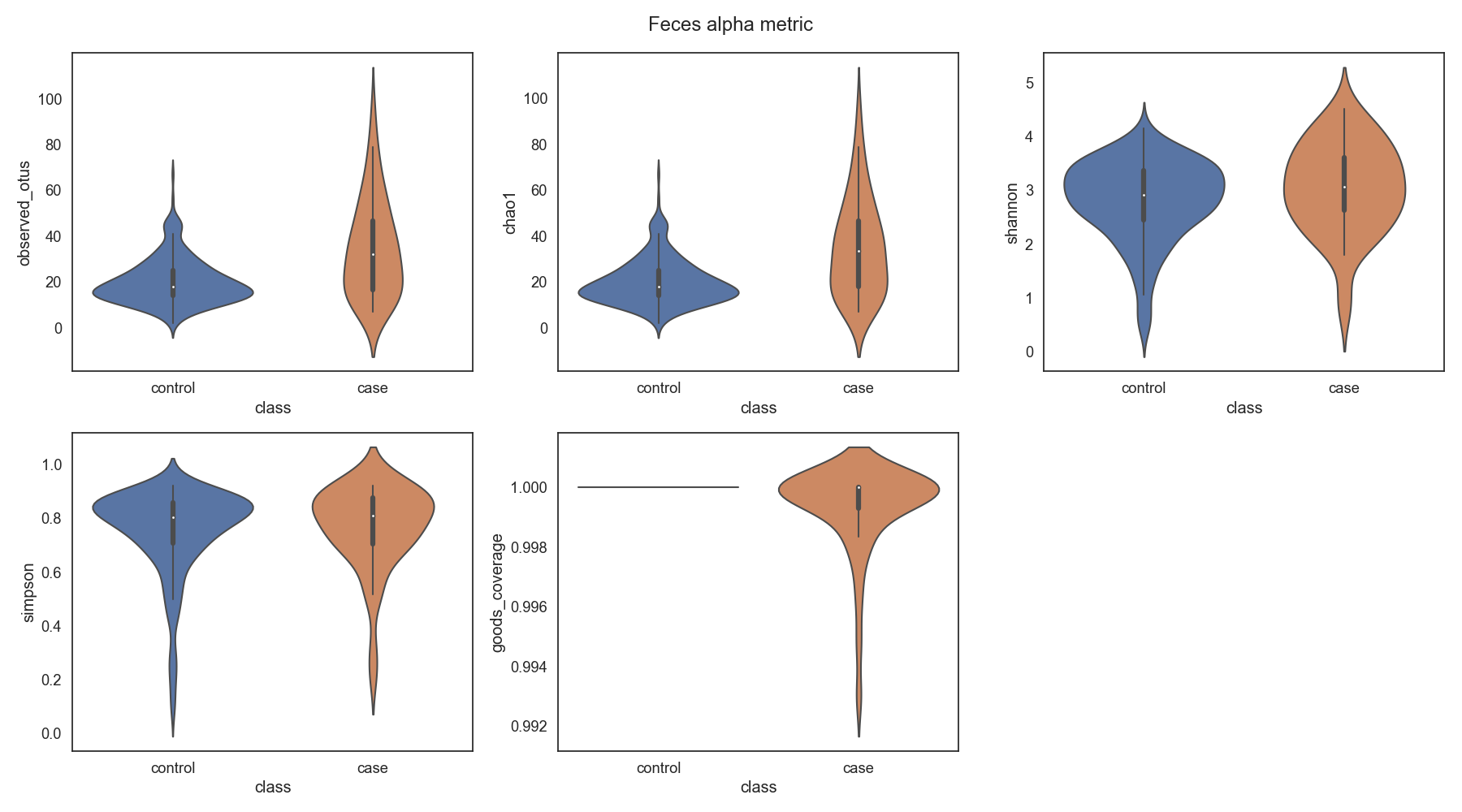
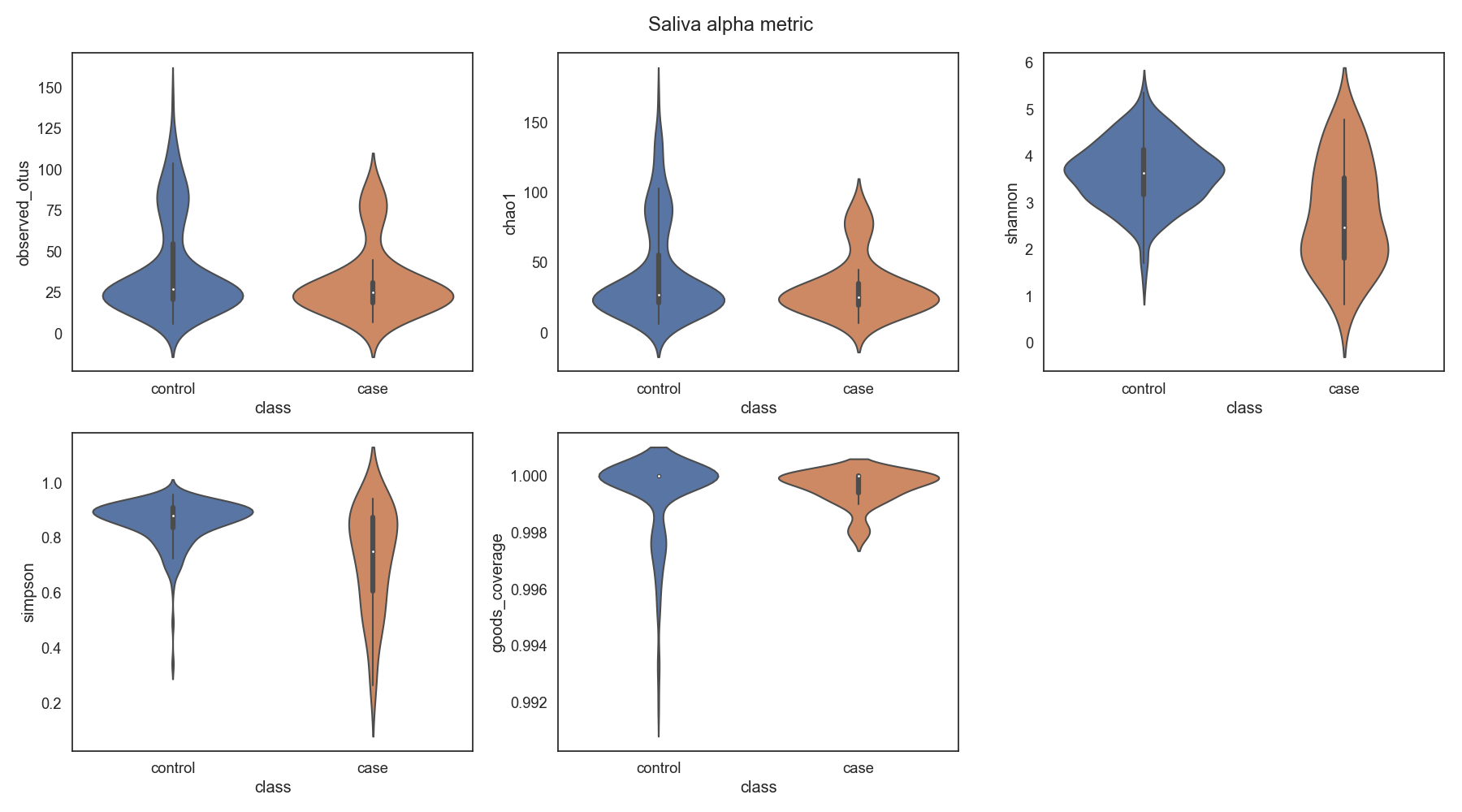
Fig19. Microbiome 다양성 분석
고지혈증 그룹 확인
헬빅쇼 데이터셋의 위암 그룹은 "RRR" 입니다. 전체 환자수에서 위암이 차지하는 비율을 파이차트로 확인합니다. 그리고, case 그룹을 위암그룹으로 하고 나머지는 control 로 하여, 비교 분석을 수행합니다.
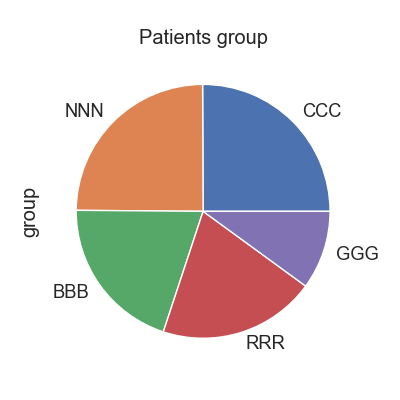
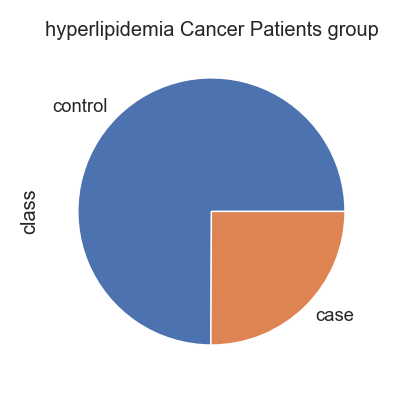
Fig1. 환자 그룹별 파이차트
변이 시그니처 분석
WGS 데이터로 부터, 변이 종류별 변이 갯수를 카운팅한 후, 이를 NMF 분석하여 7개의 시그니처를 생성합니다. case, control 각각 시그니처 분포를 확인합니다.
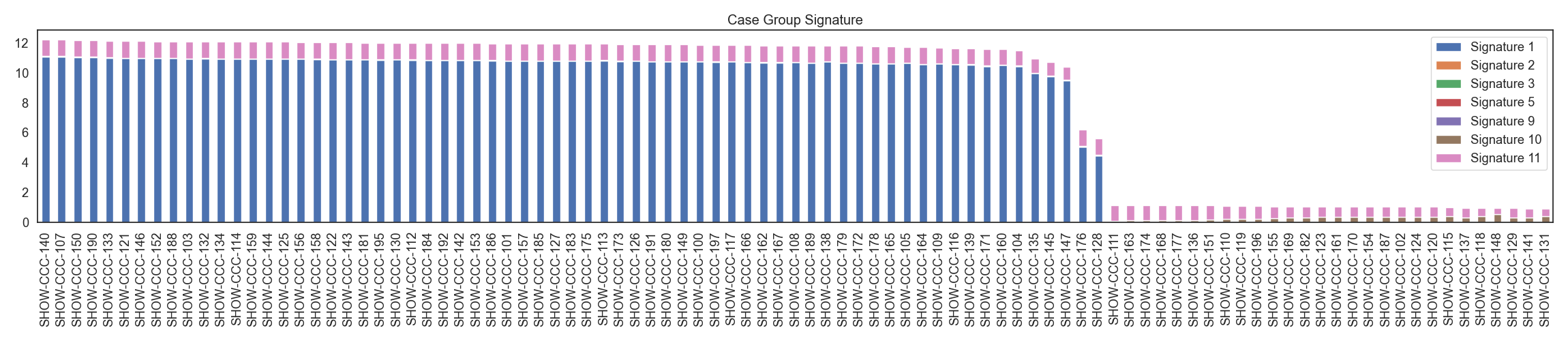
Fig2. Case 그룹 시그니처 분포
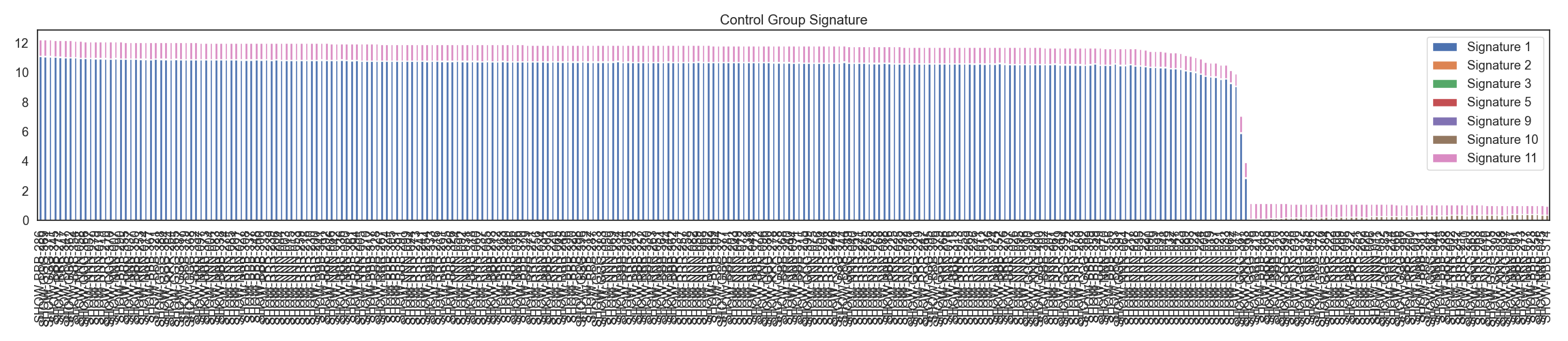
Fig3. Control 그룹 시그니처 분포
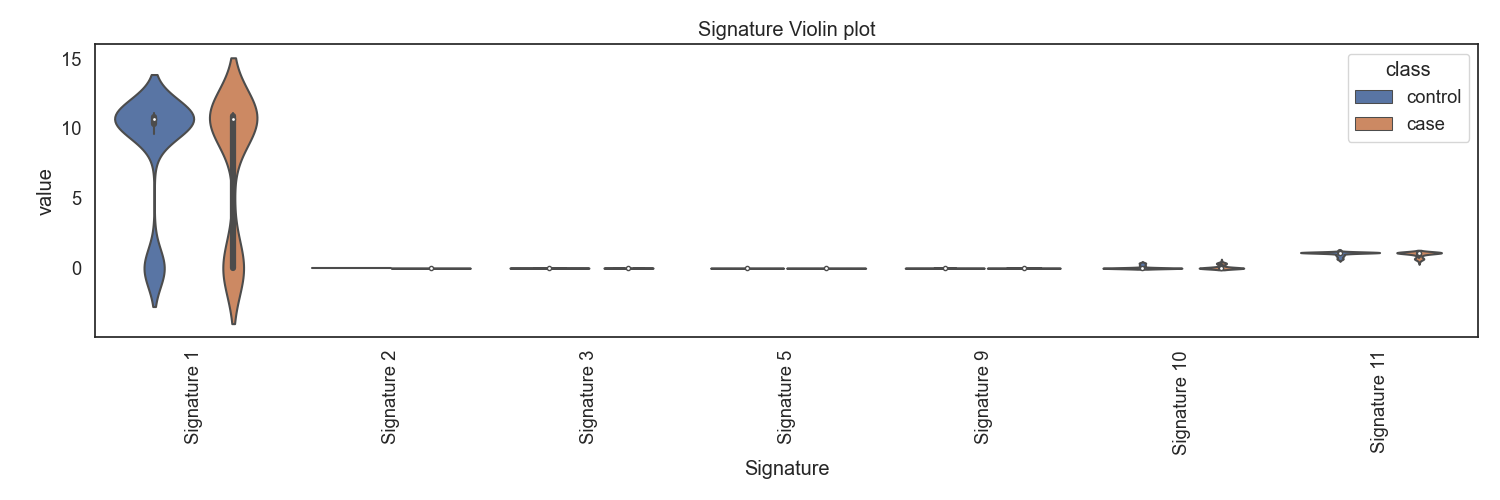
Fig4. 시그니처별 바이올린 플랏
DNA 메틸화 비율
Methyl-seq 데이터로 부터, 유전자별 DNA 메틸화 비율을 계산하고, 환자별로 평균을 계산한 후, 비교합니다.
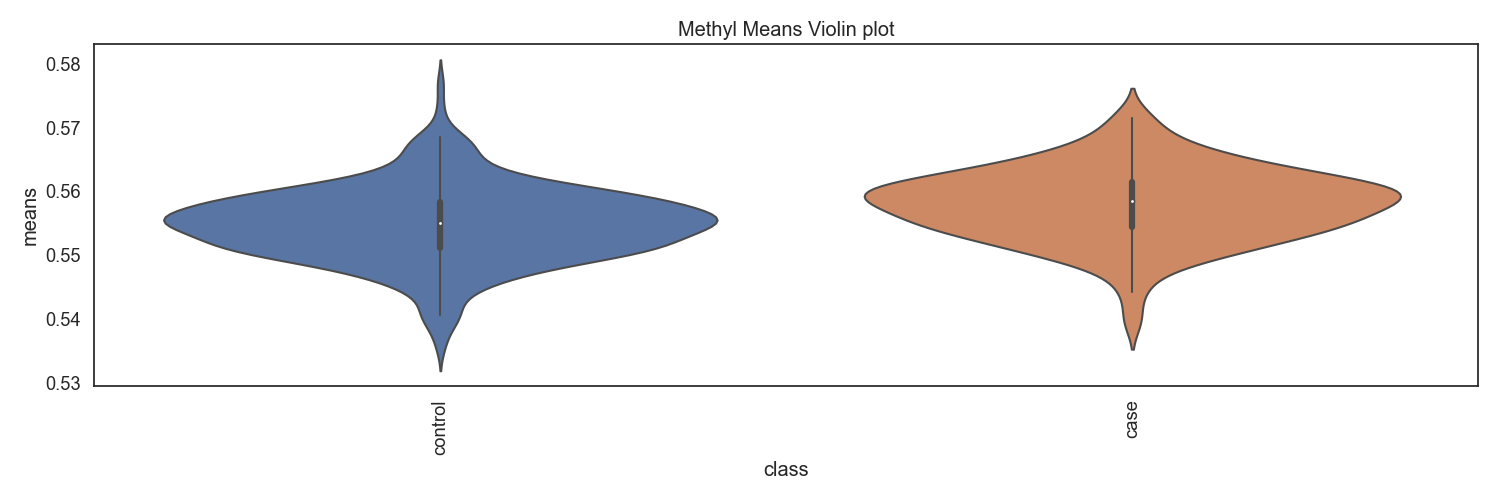
Fig5. 클래스별 메틸화 바이올린 플랏
Case/control 그룹간 차이가 큰 유전자 상위 10개의 유전자목록은 다음과 같습니다.
Fig6. 메틸화의 차가 양으로 큰 gene
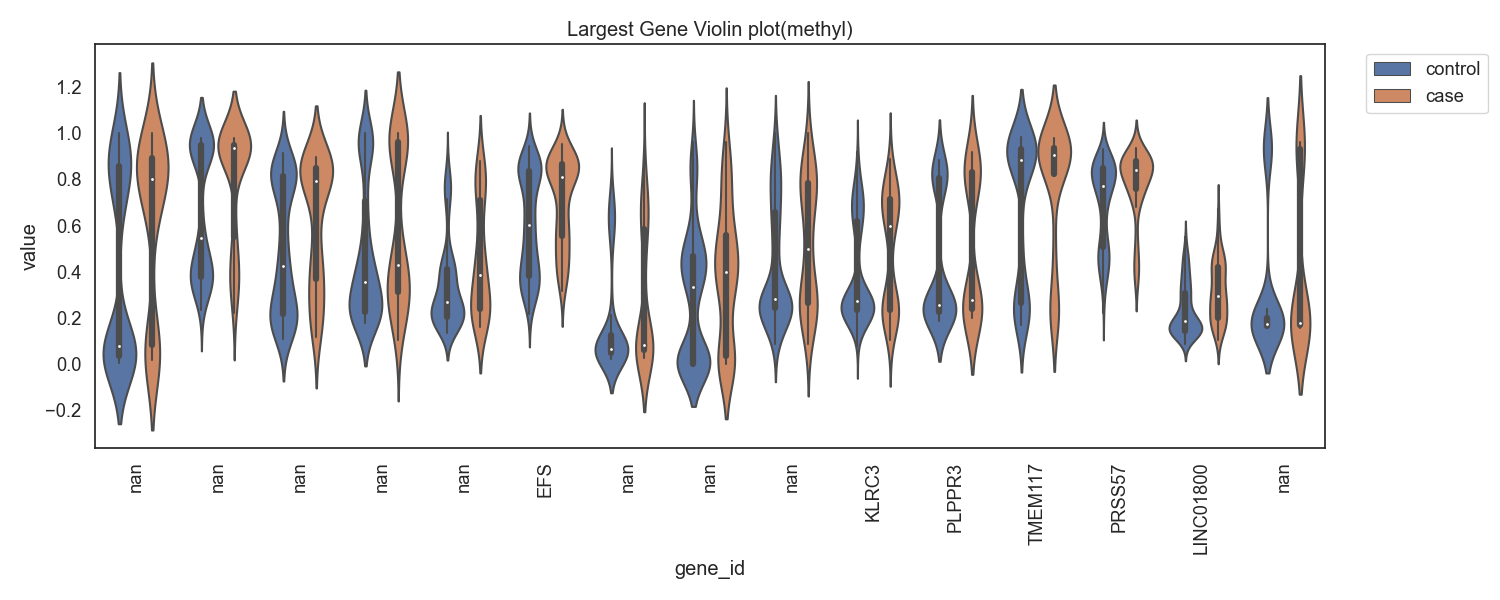
Fig7. 메틸화의 차가 양으로 큰 gene 바이올린 플랏
Fig8. 메틸화의 차가 음으로 큰 gene
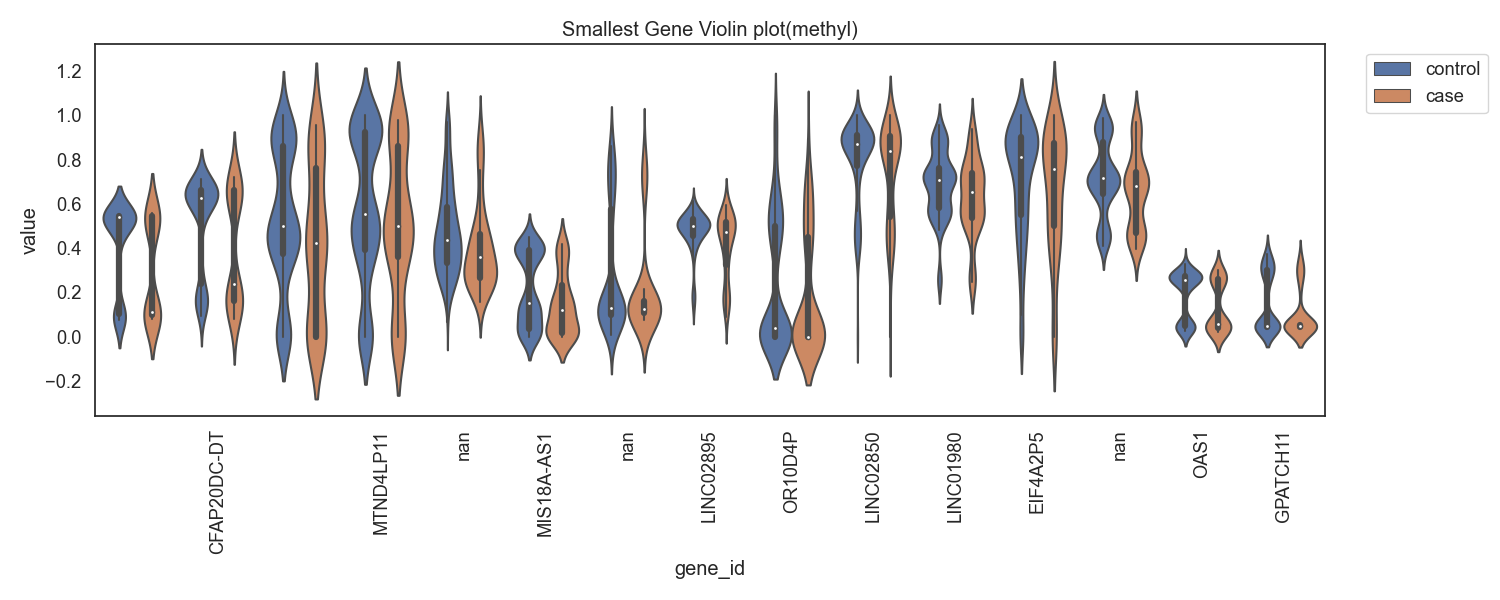
Fig9. 메틸화의 차가 음으로 큰 gene 바이올린 플랏
유전자 발현 분석
RNA-seq 분석을 통해 확인한 TPM 자료로 부터, Case/control 그룹간 전체 환자별 유전자 발현 평균 분포를 확인합니다.
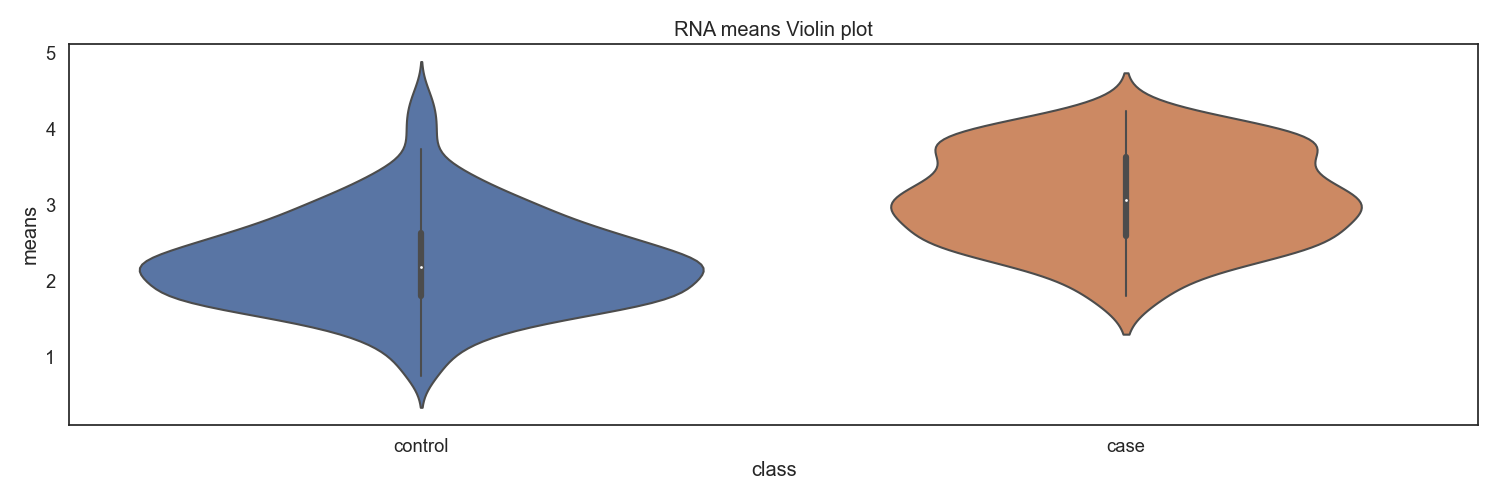
Fig10. 클래스별 RNA발현량 바이올린 플랏
Case/control 그룹간 유전자 발현 차이가 높은 유전자들의 목록을 확인합니다.
Fig11. RNA발현량의 차가 음으로 큰 gene
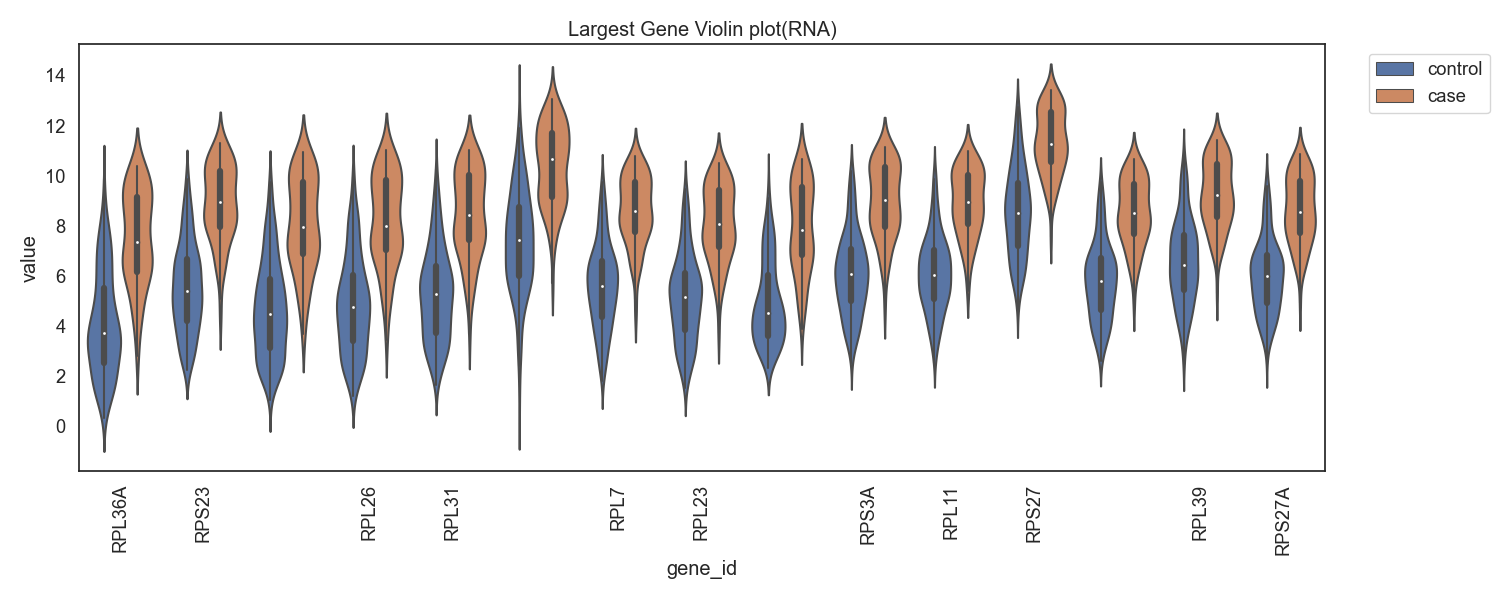
Fig12. RNA발현량의 차가 양으로 큰 gene 바이올린 플랏
Fig13. RNA발현량의 차가 음으로 큰 gene
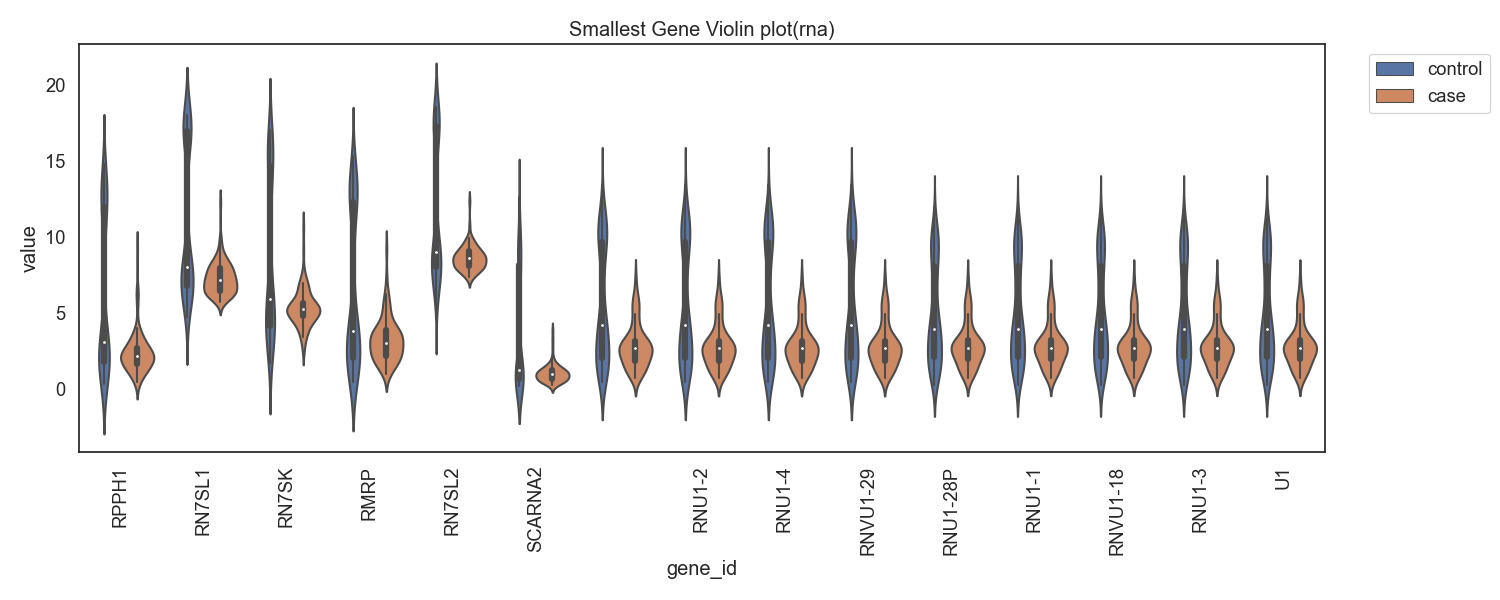
Fig14. RNA발현량의 차가 음으로 큰 gene 바이올린 플랏
DNA 메틸화와 유전자 발현과의 관계
Methyl-seq 분석 결과인 유전자별 DNA 메틸화 결과와 RNA-seq 분석 결과인 유전자별 RNA 발현 결과를 이용하여 어떠한 상관관계가 있는지 확인합니다. 유전자별 상관계수를 구하고, Case/control 그룹간 분포를 확인합니다.
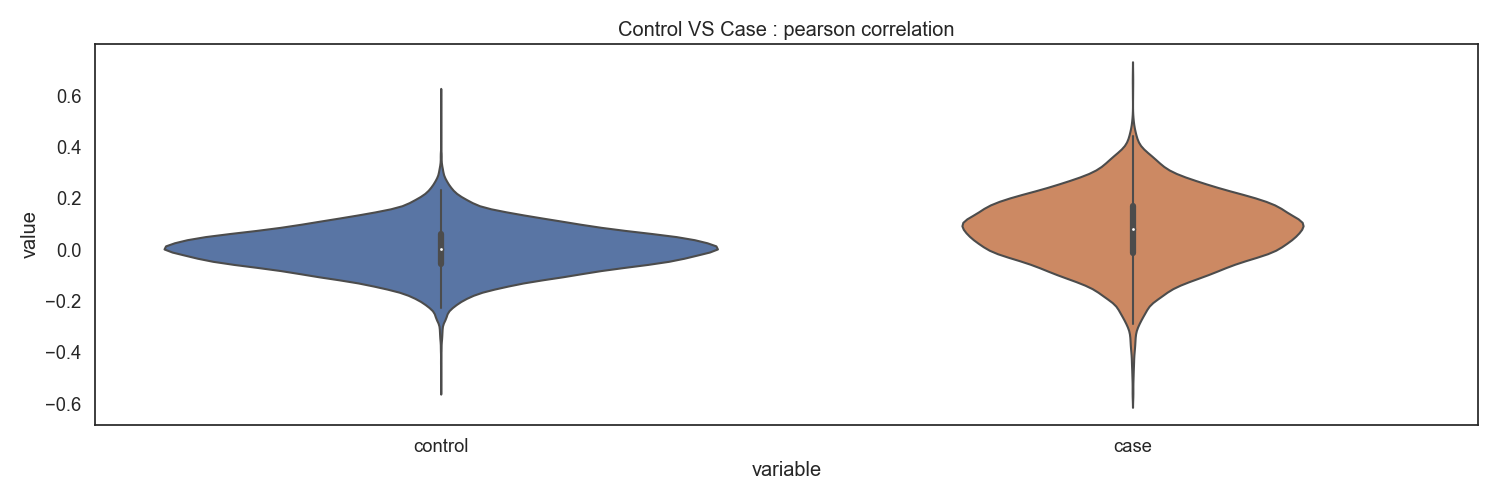
Fig15. Control vs Case : pearson correlation
DNA 메틸화와 RNA 발현량은 음의 상관관계가 있다고 알려져 있습니다. 음의 상관관계가 높은 상위 8개 유전자들을 확인합니다.
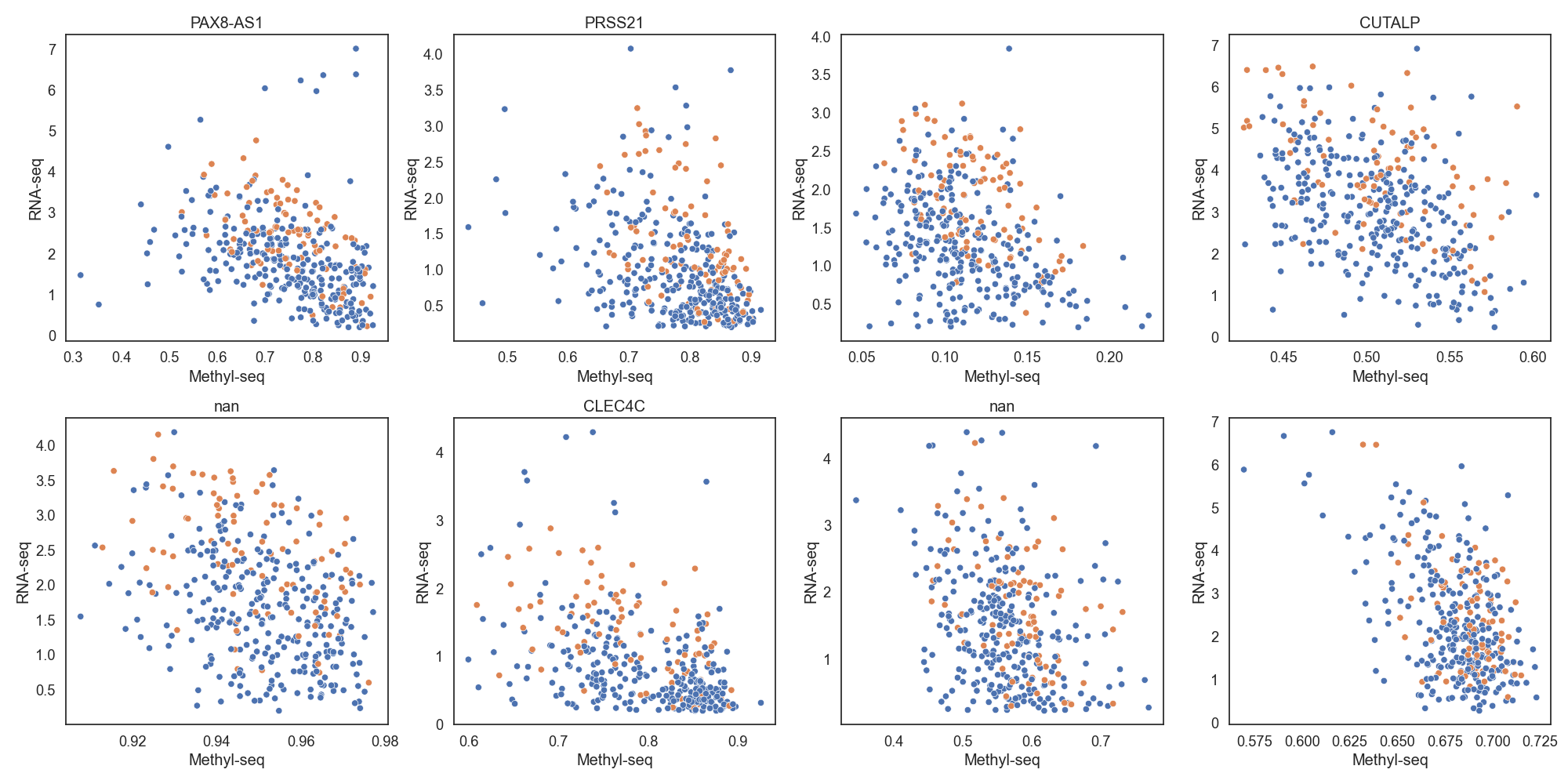
Fig16. RNA_seq, Methyl_seq scatter plot
Microbiome 분석
Case/control 각각 마이크로바이옴 풍부도 분포가 어떻게 차이나는지 확인합니다. 또한 이를 Phylum 별로 구분하여, 어떠한 미생물 계통이 더 많은지 확인합니다.
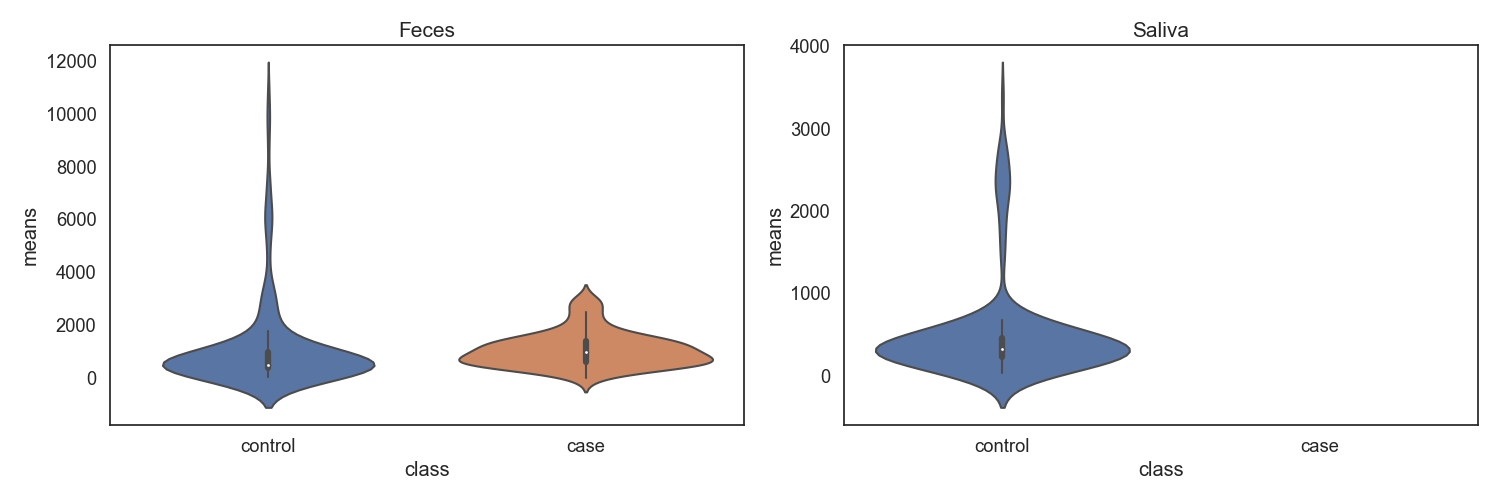
Fig17. Case vs. Control 전체 풍부도 특징
Case/control 각각 마이크로바이옴 다양성 차이가 어떠한지 확인합니다.
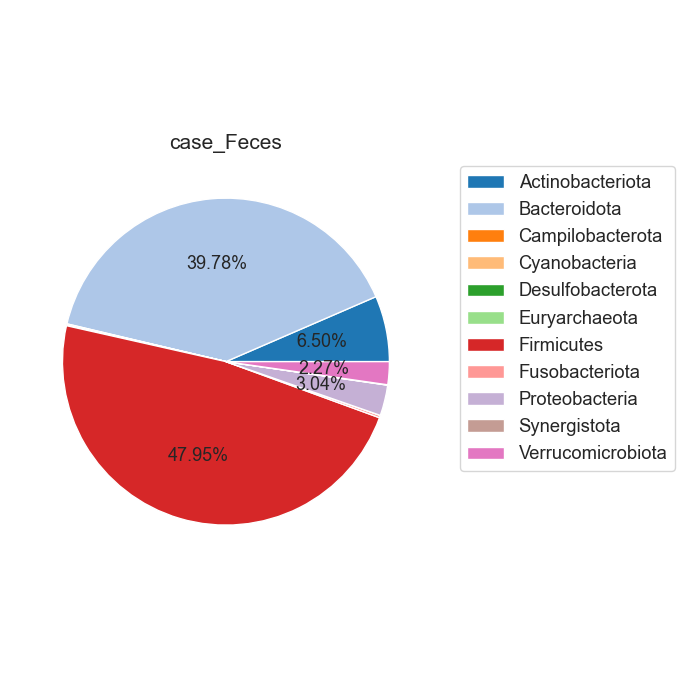
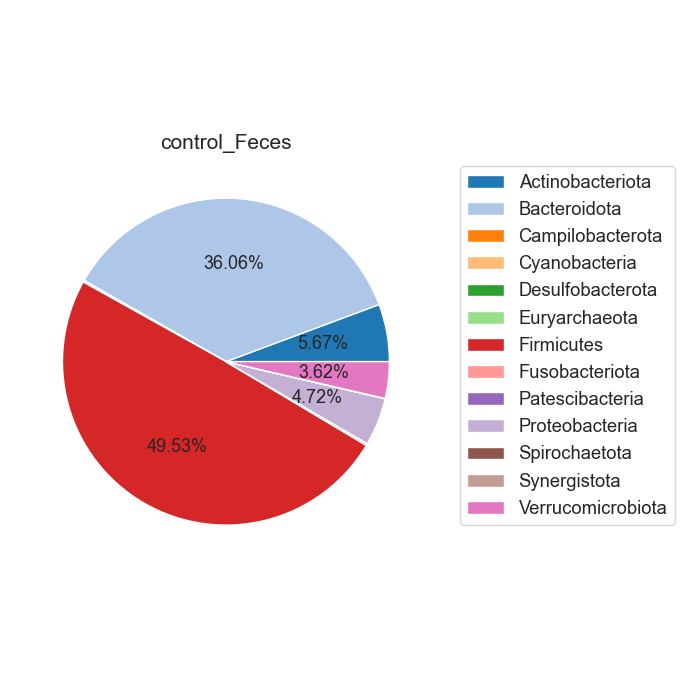
Fig18. Phylum 별 Microbiome 풍부도 특징
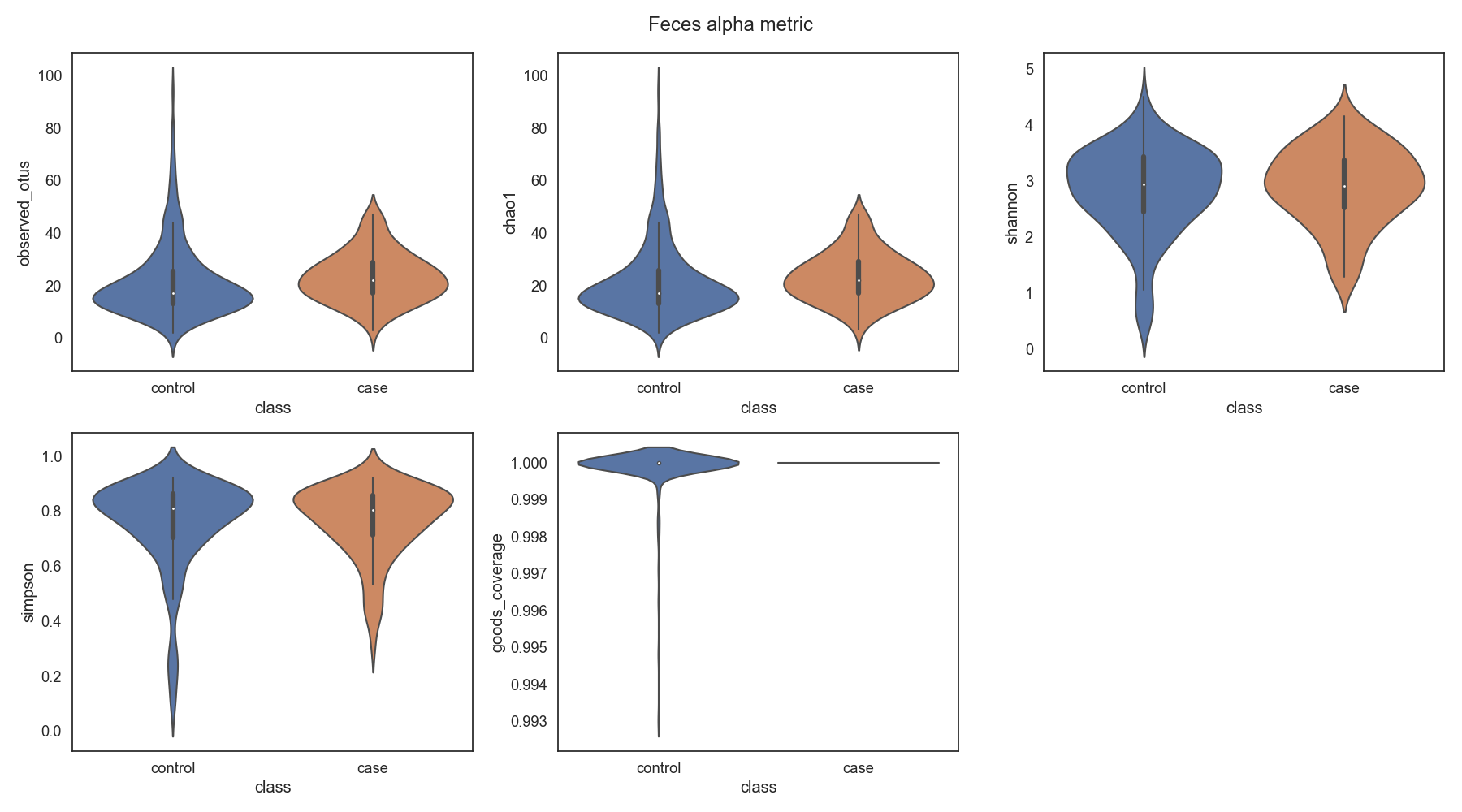
Fig19. Microbiome 다양성 분석